Sodré GB Neto
Acúmulo de Mutações Deletérias (Entropia Genética) como Fator Predominante na Prevalência Global da Intolerância à Lactose
Resumo
1. Introdução: O Paradoxo da Intolerância
O alelo LACR não silencia totalmente a lactase; ele reduz a síntese da enzima pela diminuição da transcrição do RNA mensageiro, resultando em um silenciamento parcial, de forma que tanto na vida adulta quanto na infancia haja quebra da lactose . Indivíduos homozigotos para LACR apresentam níveis muito baixos da lactase na vida adulta, mas não é um silenciamento absoluto. Já os heterozigotos LACP/LACR mantêm a produção da enzima na vida adulta, pois LACP é dominante. Portanto, o alelo LACR causa um silenciamento parcial, significativo, mas não total da lactase na fase adulta .O gene que codifica a lactase (LCT) está localizado no braço longo do cromossomo 2 e compreende 17 exons ao longo de aproximadamente 49-56 kb. Portanto, os trechos loci codificadores da lactase são os 17 exons desse gene LCT.Quanto à regulação da lactase, o gene da lactase é expresso especialmente na infância e geralmente é silenciado após o desmame (infância), o que leva à diminuição da produção da enzima na vida adulta em pessoas com o alelo restritivo (LACR). A persistência ou silenciamento da lactase na vida adulta está relacionada a alelos reguladores: o alelo LACP mantém a expressão da lactase na vida adulta, enquanto o LAC*R silencia essa expressão após a infância, reduzindo a transcrição do RNA mensageiro da lactase.Em resumo:O gene LCT possui 17 exons codificadores da lactase.A lactase é normalmente expressa na infância e silenciada na vida adulta em indivíduos com o alelo LAC*R.O silenciamento da lactase ocorre por regulação na transcrição da RNA mensageiro, não por alterações nos exons codificadores propriamente.Esses dados indicam que apenas o locus regulador, relacionado ao alelo LAC*R, é responsável pelo silenciamento da lactase na vida adulta, enquanto os 17 exons permanecem como trechos codificadores da enzima durante a vida
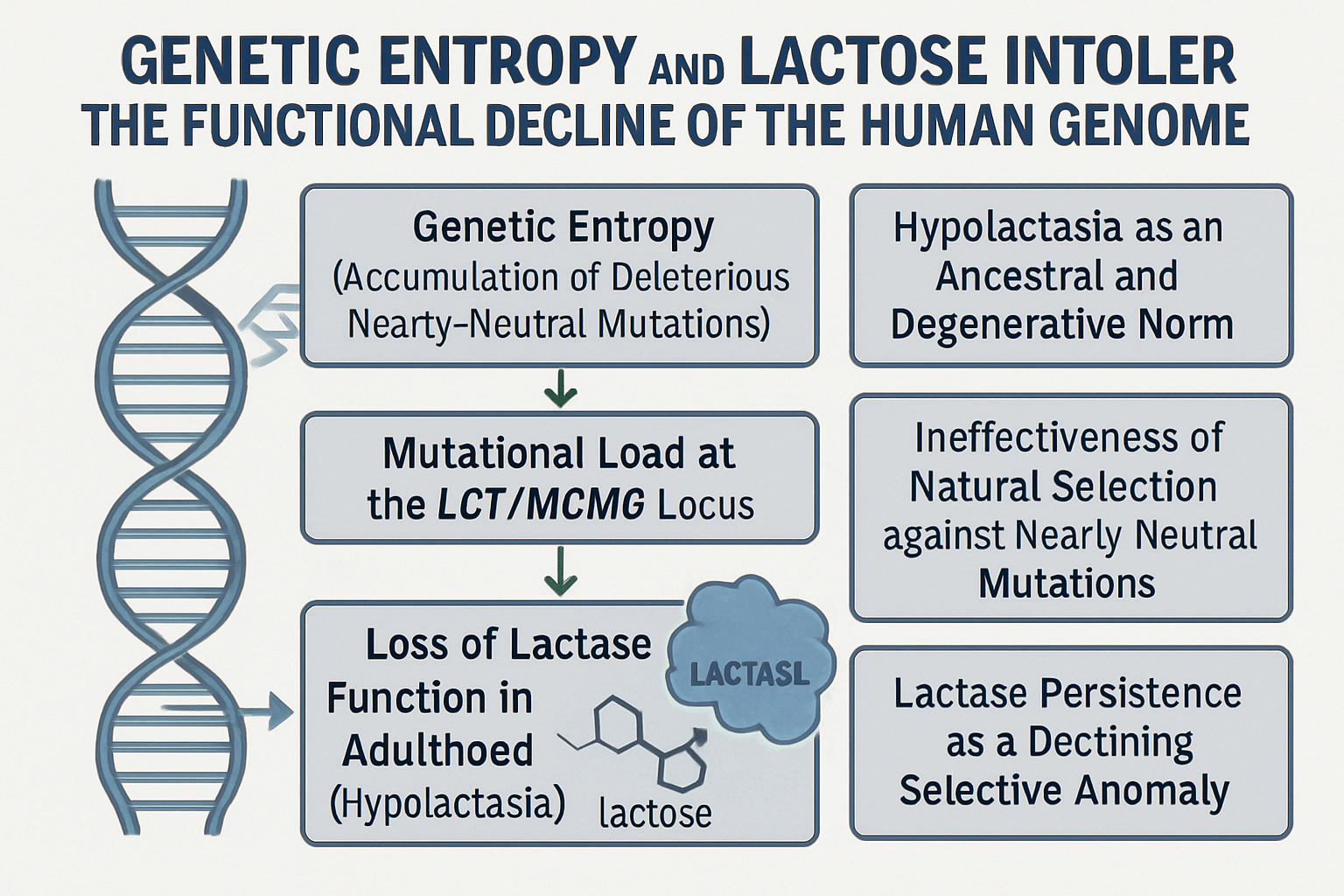
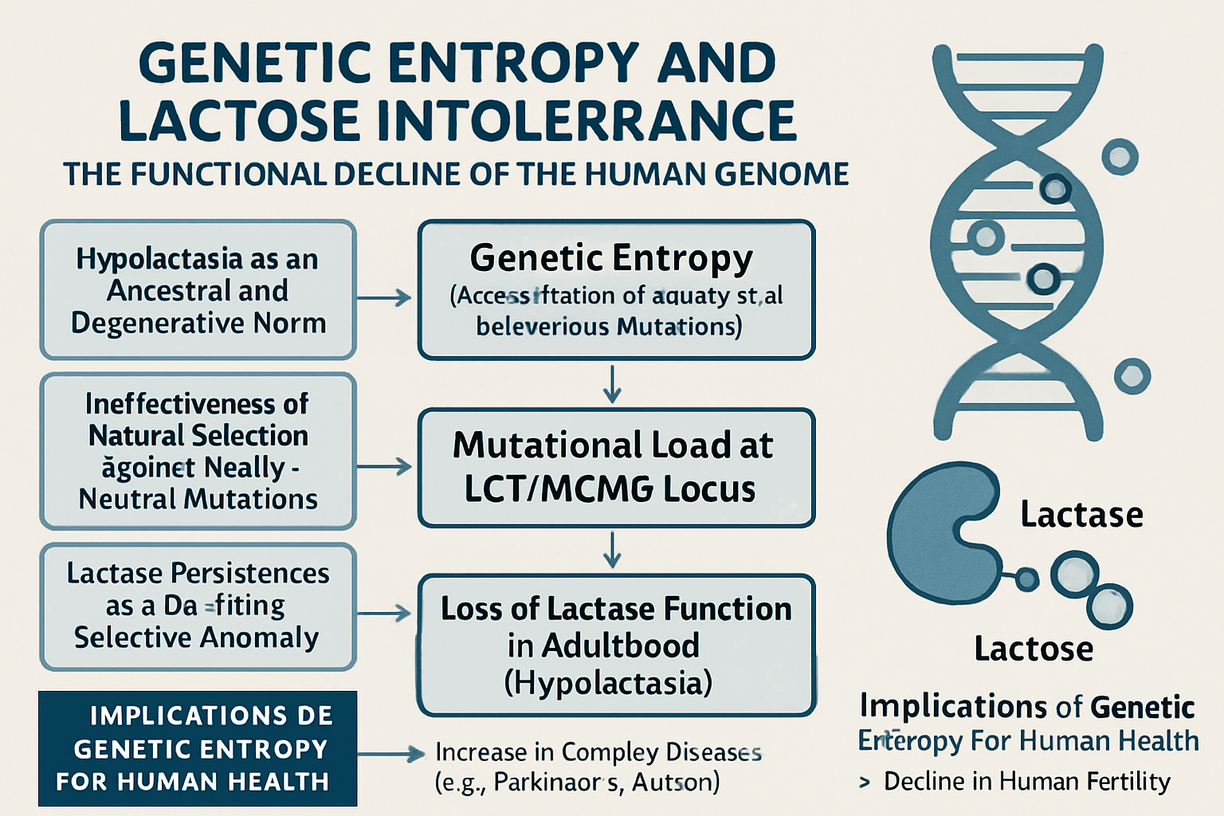
2. Entropia Genética e o Declínio Funcional
2.1. A Carga Mutacional no Locus LCT/MCM6
2.2. O Escapamento Natural e a Hipolactasia
3. Conclusão
Referências
Tabela de Artigos Científicos
|
Categoria
|
N°
|
Título do Artigo
|
Autores e Publicação
|
URL/DOI
|
|
Acúmulo de Mutações Deletérias (Carga Genética)
|
1
|
Association of the Lactase Persistence Haplotype Block With Disease Risk in Populations of European Descent
|
Joslin et al. (2020). Frontiers in Genetics.
|
|
|
2
|
Evidence for Hitchhiking of Deleterious Mutations within the Human Genome
|
Chun & Fay (2011). PLoS Genetics.
|
||
|
3
|
Genetic Signatures of Strong Recent Positive Selection at the Lactase Gene
|
Bersaglieri et al. (2004). The American Journal of Human Genetics.
|
||
|
4
|
Establishment and equilibrium levels of deleterious mutations in large populations
|
Viljoen & Erasmus (2019). Scientific Reports.
|
||
|
5
|
Uncovering the extensive trade-off between adaptive evolution and genetic load in humans
|
Tang & Li (2022). Cell Reports.
|
||
|
Perda de Lactase (Não-Persistência/Intolerância)
|
6
|
Genetics of lactase persistence and lactose intolerance
|
Swallow (2003). Annual Review of Genetics.
|
|
|
7
|
Genetics of Lactose Intolerance: An Updated Review and Online Interactive World Maps of Phenotype and Genotype Frequencies
|
Anguita-Ruiz et al. (2020). Nutrients.
|
||
|
8
|
Lactase non-persistence is directed by DNA variation-dependent epigenetic changes in the LCT enhancer
|
Labrie et al. (2016). Nature Structural & Molecular Biology.
|
||
|
9
|
Differences in DNA Methylation and Functional Expression in Lactase Persistent and Non-Persistent Individuals
|
Leseva et al. (2018). Scientific Reports.
|
||
|
10
|
Four novel mutations in the lactase gene (LCT) underlying congenital lactase deficiency in patients with different ethnic origins
|
Torniainen et al. (2009). BMC Gastroenterology.
|
||
|
11
|
Mutations in the Translated Region of the Lactase Gene (LCT) Underlie Congenital Lactase Deficiency
|
Kuokkanen et al. (2006). The American Journal of Human Genetics.
|
||
|
12
|
Changing genes; losing lactase
|
Grand et al. (2003). Gut.
|
||
|
13
|
Lactase Non-persistence and Lactose Intolerance
|
Bayless & Rosensweig (2017). Gastrointestinal and Liver Disease.
|
||
|
Contexto Evolutivo e Implicações
|
14
|
Evolution of lactase persistence: an example of human niche construction
|
Gerbault et al. (2011). Philosophical Transactions of the Royal Society B.
|
|
|
15
|
Dairying and the evolution and consequences of lactase persistence
|
Stock & Thomas (2023). Annual Review of Food Science and Technology.
|
||
|
16
|
Evolution of Lactase Persistence: Turbo-Charging Adaptation and Its Implications for Contemporary Health
|
Wells (2021). Frontiers in Nutrition.
|
||
|
17
|
On the Evolution of Lactase Persistence in Humans
|
Ségurel & Bon (2017). Annual Review of Genomics and Human Genetics.
|
||
|
18
|
Lactase persistence versus lactose intolerance: Is there an evolutionary advantage?
|
Dzialanski & Sibley (2016). Current Opinion in Gastroenterology.
|
||
|
19
|
Evolutionary and molecular genetics of regulatory alleles underlying lactase persistence in humans
|
Liebert (2014). Tese de Doutorado.
|
||
|
20
|
Milk, mortality, and genetics
|
Duarte & Thomas (2014). BMJ.
|
Referências
Perda da Capacidade de Produzir Lactase (Não-Persistência da Lactase – LNP)
Artigos Focados na Perda de Lactase (Não-Persistência/Intolerância)
|
N°
|
Título do Artigo
|
Autores e Publicação
|
Foco Principal
|
URL/DOI
|
|
1
|
Genetics of lactase persistence and lactose intolerance
|
Swallow, D. M. (2003). Annual Review of Genetics.
|
Revisão clássica que define a LNP como o estado ancestral e discute a genética da persistência e não-persistência da lactase.
|
|
|
2
|
Genetics of Lactose Intolerance: An Updated Review and Online Interactive World Maps of Phenotype and Genotype Frequencies
|
Anguita-Ruiz et al. (2020). Nutrients.
|
Revisão atualizada sobre a genética da intolerância à lactose, detalhando os mecanismos da LNP e LP.
|
|
|
3
|
Lactase non-persistence is directed by DNA variation-dependent epigenetic changes in the LCT enhancer
|
Labrie et al. (2016). Nature Structural & Molecular Biology.
|
Explora o mecanismo molecular da LNP, mostrando que ela é dirigida por mudanças epigenéticas (metilação do DNA) no enhancer do gene LCT.
|
|
|
4
|
Differences in DNA Methylation and Functional Expression in Lactase Persistent and Non-Persistent Individuals
|
Leseva et al. (2018). Scientific Reports.
|
Corrobora o papel da metilação do DNA na regulação da expressão do gene LCT e, consequentemente, na perda de lactase (LNP).
|
|
|
5
|
Four novel mutations in the lactase gene (LCT) underlying congenital lactase deficiency in patients with different ethnic origins
|
Torniainen et al. (2009). BMC Gastroenterology.
|
Detalha mutações específicas que causam a Deficiência Congênita de Lactase (CLD), uma forma severa e precoce de perda de lactase.
|
|
|
6
|
Mutations in the Translated Region of the Lactase Gene (LCT) Underlie Congenital Lactase Deficiency
|
Kuokkanen et al. (2006). The American Journal of Human Genetics.
|
Foca nas mutações deletérias raras que causam a Deficiência Congênita de Lactase (CLD).
|
|
|
7
|
Changing genes; losing lactase
|
Grand et al. (2003). Gut.
|
Artigo que discute a base genética da persistência da lactase e a perda de lactase (LNP) como a norma na maioria dos adultos.
|
|
|
8
|
Lactase Non-persistence and Lactose Intolerance
|
Bayless & Rosensweig (2017). Gastrointestinal and Liver Disease.
|
Capítulo de livro que define a LNP como a condição genética onde a atividade da lactase é baixa, levando à intolerância à lactose.
|
Referências
Genética da Intolerância à Lactose: Uma Revisão Atualizada com Mapas Interativos Mundiais
Uma análise abrangente dos fenótipos e genótipos de persistência e não-persistência da lactase em populações humanas, com ênfase nos mecanismos moleculares e evolutivos que levam à perda da capacidade de digerir lactose na idade adulta.
Introdução: A Lactose como Açúcar Único nos Mamíferos
Estrutura Molecular
A lactose (β-d-Galactopiranosil-(1→4)-d-glucose) é o principal carboidrato presente no leite, constituindo uma fonte essencial de energia durante o período de amamentação. Este dissacarídeo requer hidrólise no trato intestinal para que seus monossacarídeos componentes, galactose e glicose, possam ser absorvidos pelos enterócitos.
A concentração de lactose varia entre espécies de mamíferos: enquanto o leite humano contém aproximadamente 7g por 100mL, outros mamíferos apresentam concentrações menores (4,8g em vacas, 4,1g em cabras e 4,8g em ovelhas).
Degradação Enzimática
A hidrólise da lactose no intestino é catalizada pela enzima lactase-florizina hidrolase (LPH), uma β-galactosidase localizada na membrana da borda em escova dos enterócitos do intestino delgado. Esta enzima é expressa exclusivamente no intestino delgado humano, sendo confinada aos enterócitos absortivos nas vilosidades.
A atividade da LPH demonstra regulação posicional ao longo do eixo proximal-distal, com níveis elevados no meio do jejuno e níveis reduzidos no duodeno e íleo distal, padrão que se assemelha ao de outra hidrolase digestiva, a sacarase-isomaltase.
A Enzima Lactase-Florizina Hidrolase: Estrutura e Função
Estrutura Proteica
Glicoproteína transmembranar de 160 kDa
C-terminal intracelular de 26 aminoácidos
N-terminal na superfície luminal
Região transmembranar hidrofóbica de 19 resíduos
Domínios Funcionais
Homologia interna quádrupla (domínios I-IV)
Domínios I e II: regulação do dobramento proteico
Domínios III e IV: glicosilados e com sítios ativos
Cada sítio ativo contém um resíduo de glutamato nucleófilo
Processamento Pós-Traducional
LPH nascente: 195 kDa no retículo endoplasmático
Glicosilação dependente de dolicol: 215 kDa
Glicosilação complexa no Golgi: 220 kDa
Clivagem extracelular produz enzima madura funcional
A enzima LPH é multifuncional, capaz de hidrolisar não apenas lactose, mas também lactosilceramida, celobiose, celotriose, glicosídeos flavonoides e florizina. Esta versatilidade enzimática sugere uma função metabólica mais ampla além da simples digestão da lactose do leite.
O Gene LCT: Localização e Organização Genômica
O gene humano LCT está localizado no cromossomo 2q21, compreendendo 17 éxons e abrangendo aproximadamente 49 kb de DNA genômico. O gene codifica um RNA mensageiro de pouco mais de 6 kb que, do códon de iniciação ao códon de parada, traduz 1927 aminoácidos formando o produto completo da tradução.
Elementos Regulatórios Proximais
Os primeiros 100 bp do promotor proximal de LCT são virtualmente idênticos entre mamíferos analisados (rato, camundongo, porco e humano). Esta região contém sítios de ligação para fatores de transcrição críticos:
- Cdx-2 (caudal type homeobox 2)
- HNF1-α (hepatocyte nuclear factor 1α)
- Fatores de transcrição GATA
- HOXC11, HNF-3, C/EBP e FREAC-2/3
Características Únicas Humanas
Diferentemente de outros mamíferos, os primeiros 1000 bp da região 5′-flanqueadora do gene LCT humano contém duas sequências Alu de aproximadamente 300 bp cada. A região mais distal contém sequências adicionais de DNA repetitivo, dificultando a comparação direta com regiões regulatórias de outros mamíferos.
O impacto funcional destes segmentos de DNA repetitivo inseridos na expressão de LCT ainda permanece desconhecido.
O Enhancer MCM6: Elemento Regulatório Crítico
O éxon 17 do gene MCM6 (minichromosome maintenance complex component 6), um gene regulador do ciclo celular que termina 3,5 kb antes do sítio de início do gene LCT humano, desempenha papel crucial como enhancer regulatório transcricional de LCT. O sítio de início transcricional do gene MCM6 localiza-se aproximadamente 39 kb 5′ do sítio de início transcricional de LCT.
Proximidade Genômica
MCM6 e LCT são genes próximos no cromossomo 2q21, mas as evidências disponíveis indicam que sua regulação é independente. Esta proximidade permitiu a evolução de elementos regulatórios no MCM6 que afetam especificamente a expressão de LCT.
Função de Enhancer
A região intrônica do MCM6 contém elementos regulatórios que podem aumentar ou suprimir a transcrição do gene LCT a distâncias consideráveis. Mutações nesta região são responsáveis pela persistência da lactase em adultos.
Sítio de Mutações
Polimorfismos de nucleotídeo único (SNPs) localizados no enhancer MCM6 criam ou destroem sítios de ligação para fatores de transcrição, modulando assim a expressão de LCT ao longo da vida.
Fenótipos da Lactase: Persistência versus Não-Persistência
Não-Persistência da Lactase (LNP)
Prevalência: ~70% da população adulta mundial
Característica: Declínio programado geneticamente da atividade da lactase após o desmame, reduzindo para <10% dos valores neonatais
Manifestação: Capacidade reduzida de digerir lactose (LDC), frequentemente levando a sintomas após consumo de leite fresco
Persistência da Lactase (LP)
Prevalência: ~30% da população adulta mundial
Característica: Manutenção de níveis elevados de atividade da lactase ao longo da vida adulta
Manifestação: Capacidade de hidrolisar grandes quantidades de lactose e consumir grandes quantidades de leite fresco sem complicações
É fundamental diferenciar a não-persistência da lactase (hipolactasia tipo adulto ou restrição de lactase) de outras condições. A deficiência congênita de lactase é extremamente rara e aparece no início da amamentação, causada por mutações graves em ambos os alelos do gene LCT. A hipolactasia secundária é causada por dano direto à lactase da mucosa (por exemplo, celíaca, giardíase, enterite) e não tem base genética.
Terminologia Clínica Importante: Má absorção de lactose refere-se à diminuição da elevação da glicose sanguínea ou aumento da elevação do hidrogênio no ar expirado após uma carga oral de lactose. Intolerância à lactose descreve os sintomas que normalmente acompanham a má absorção de lactose (diarreia, flatulência, dor abdominal).
Base Genética da Não-Persistência: Mecanismos de Repressão
Declínio Natural Programado da Lactase
Embora consideráveis esforços de pesquisa tenham sido dedicados à compreensão dos mecanismos genéticos subjacentes à LNP, algumas lacunas permanecem não preenchidas. O declínio natural da lactase parece resultar da ação direta de repressores transcricionais, aproximadamente aos 5 anos de idade, que provocam a redução dos níveis de lactase.
Repressão Transcricional
Segundo estudos de promotor, um candidato plausível é o fator de transcrição Pdx-1, que demonstrou regulação negativa direta da expressão de LCT in vitro. A ação de repressores transcricionais leva à diminuição da taxa de transcrição de LPH.
Redução da Síntese Proteica
A diminuição da transcrição provoca uma consequente redução na síntese da proteína LPH e da atividade enzimática ao longo do gradiente proximal-distal do intestino delgado, bem como a virtual extinção da expressão no duodeno e íleo distal.
Mecanismos Epigenéticos
Além da ação de fatores de transcrição, alguns mecanismos epigenéticos (principalmente metilação do DNA) também podem estar envolvidos na regulação da LNP, como demonstrado por Labrie et al. (2016).
Modificações Pós-Traducionais
Diferenças interpessoais na glicosilação terminal das cadeias laterais de carboidratos da lactase, devido a polimorfismos nos genes Lewis (FUT3) e Secretor (FUT2), influenciam a estabilidade proteica e podem contribuir para o declínio natural dos níveis intestinais de lactase.
Metilação do DNA e Não-Persistência da Lactase
Descobertas Epigenéticas Fundamentais
Um marco importante na compreensão da LNP foi o trabalho de Labrie et al. (2016), que demonstrou que o alelo C da variante -13910:C>T está associado ao aparecimento e acúmulo de metilações relacionadas à idade tanto nas regiões MCM6 quanto LCT.
Estes autores comprovaram que estas metilações dependentes da variação do DNA provocam silenciamento da expressão de LCT, sendo assim um contribuinte plausível para a LNP em mamíferos. Esta descoberta revolucionou a compreensão dos mecanismos moleculares da intolerância à lactose.
Em linha com as descobertas de Labrie, outro trabalho demonstrou como certas assinaturas epigenéticas, em elementos enhancer e promotor de LCT, alinham-se com a variação transcricional de LCT em enterócitos de camundongos.
Impacto da Exposição à Lactose
Estudos revelaram que pistas ambientais específicas da idade e do fenótipo (exposição à lactose após o desmame) induzem mudanças nessas modificações epigenéticas e alteram o gradiente de mRNA de LCT intestinal.
Um estudo recente identificou meQTLs (loci de traços quantitativos de metilação) putativos de LCT, que são diferencialmente metilados entre indivíduos LP e LNP. Notavelmente, metilações de DNA nos sítios enhancer e promotor de LCT, ao invés da expressão de fatores de transcrição intestinais como CDX2, POU2F1, GATA4/6 ou HNF1-α, foram preditivas de LNP/LP.
Implicação Clínica: A variação de metilação do DNA apresentou maior poder para prever fenótipos de lactase do que os alelos LP clássicos isoladamente, sugerindo um papel plausível da epigenética no aparecimento e regulação dos fenótipos de lactase.
Mecanismos Genéticos Subjacentes aos Fenótipos de Lactase
Este diagrama ilustra de forma abrangente os mecanismos genéticos que fundamentam a persistência da lactose (LP) e a não-persistência da lactase (LNP) em humanos. As formas retangulares representam todos os fatores de transcrição atualmente conhecidos que interagem com o promotor do gene da lactase (LCT).
Ativadores Transcricionais
CDX-2, HNF1-α, GATA e OCT-1 são conhecidos por promover a expressão de LCT. Estes fatores de transcrição ligam-se a elementos regulatórios específicos na região promotora do gene, aumentando a taxa de transcrição e, consequentemente, a produção da enzima lactase.
Repressor Transcricional
O PDX-1 foi descrito como um repressor transcricional que diminui ativamente a expressão de LCT. Este fator de transcrição liga-se ao promotor de LCT e inibe sua transcrição, contribuindo para o declínio natural da lactase após o desmame.
Modificações Epigenéticas
As formas ovais (nomeadas como “CH3”) referem-se ao aparecimento de metilações dentro da região LCT, que também foram descritas como reprimindo a expressão de LCT. A metilação do DNA é um mecanismo epigenético crucial que pode silenciar genes sem alterar a sequência de DNA.
Alelos de Persistência
Os diferentes alelos associados à LP descritos em MCM6, responsáveis pela ligação ao OCT-1, estão representados em laranja. Estas variantes genéticas criam novos sítios de ligação para fatores de transcrição, permitindo a expressão contínua de LCT na idade adulta.
Base Genética da Persistência da Lactase
Por outro lado, a persistência da atividade de LPH ao longo da idade adulta (LP) parece ser um traço genético que segue um padrão de herança dominante, envolvendo a ação de mutações de elementos cis. Particularmente, foi descrito que o efeito de vários SNPs leva a um caminho alternativo para a expressão de LCT que não é regulado negativamente, como o caminho original é.
2002: Primeira Mutação LP Descoberta
A variante -13910:C>T (rs4988235) foi identificada por Enattah et al. em um estudo de famílias finlandesas. Esta descoberta marcou o início da compreensão molecular da persistência da lactase, quase um século após o fenótipo ter sido descrito.
2007: Mutações Africanas Identificadas
Tishkoff et al. descobriram quatro variantes adicionais associadas à LP em populações africanas: -13907:C>G, -13915:T>G, -14009:T>G e -14010:G>C. Esta descoberta revolucionou a compreensão da evolução convergente da LP.
2009-2020: Expansão do Conhecimento
Dezoito novos SNPs mapeando o MCM6 foram associados à LP em populações específicas, totalizando vinte e três SNPs conhecidos que atualmente fundamentam a etiologia genética da LP.
Mecanismo Molecular Atual
O mecanismo molecular para estes SNPs é a criação de novos sítios de ligação para fatores de transcrição específicos, como a proteína de ligação ao octâmero 1 (Oct-1), que permite expressão contínua de LCT.
As 23 Variantes Genéticas Associadas à Persistência da Lactase
Até o momento, vinte e três polimorfismos de nucleotídeo único (SNPs) dentro da região regulatória MCM6 foram associados à persistência da lactase em populações humanas. Estas variantes parecem ter surgido durante o mesmo período, mas independentemente em diferentes populações humanas, razão pela qual a LP tornou-se um exemplo clássico de evolução regulatória convergente e co-evolução gene-cultura.
| SNP | RS-id | Informações Adicionais |
| -14010:G>C | rs145946881 | Amplamente estudada e associada – validada funcionalmente |
| -14009:T>G | rs869051967 | Amplamente estudada e associada – validada funcionalmente |
| -13915:T>G | rs41380347 | Amplamente estudada e associada – validada funcionalmente |
| -13910:C>T | rs4988235 | Amplamente estudada e associada – validada funcionalmente |
| -13907:C>G | rs41525747 | Amplamente estudada e associada – validada funcionalmente |
| -22018:G>A | rs182549 | Em desequilíbrio de ligação completo com -14010:G>C causal |
| -14011:C>T | rs4988233 | Alguma evidência para papel funcional |
| -13779:G>C | rs527991977 | Alguma evidência para papel funcional |
| -13730:T>G | rs4954492 | Variante frequente, em estudo |
| -13603:C>T | rs56348046 | Variante frequente, em estudo |
| -13495:C>T | rs4954490 | Variante frequente, em estudo |
Das vinte e três variantes genéticas incluídas, existem dez com frequência rara (q < 5%) nas populações onde foram descobertas, bem como outro SNP em desequilíbrio de ligação completo com o bem conhecido -13910:C>T. Para os doze SNPs frequentes e amplamente estudados restantes, bancos de dados abrangentes de frequências alélicas mundiais foram compilados.
Metodologia de Diagnóstico dos Fenótipos de Lactase
Biópsia Intestinal
Método direto e mais preciso: determinação direta da atividade da lactase a partir de uma biópsia do intestino delgado. Embora seja o padrão-ouro, constitui uma técnica invasiva e impraticável para todas as situações clínicas.
Teste de Glicose Sanguínea
Método indireto: medição do aumento da glicose no sangue após administração de uma carga de lactose de 50g. Pessoas com alta atividade de lactase apresentarão aumento significativo na concentração de glicose sanguínea dentro de 15 a 45 minutos após administração da lactose.
Teste de Hidrogênio Expirado
Método preferido: teste do hidrogênio expirado após uma carga de lactose ou, idealmente, após ingestão de uma porção (250 mL) de leite pasteurizado ou esterilizado. Em pessoas LNP, lactose não digerida alcança o cólon onde é fermentada, levando à produção de ácidos graxos e gases, incluindo hidrogênio.
Testes Genéticos
Vários testes genéticos tornaram-se disponíveis como alternativas aos testes de tolerância à lactose e biópsias intestinais. Embora constituam ferramentas diagnósticas não invasivas e úteis, dependendo das variantes genéticas analisadas, nem sempre são apropriados para todas as populações.
Distribuição Mundial dos Fenótipos de Lactase
Um impressionante número de estudos desde a década de 1960 investigou a distribuição de fenótipos de lactase em todo o mundo, relatando frequências de LP acentuadamente diferentes entre populações humanas distintas. Este mapa interativo constitui o recurso mais abrangente e atualizado para explorar as frequências de LP em todo o mundo, incluindo todos os relatos de literatura sobre frequências de LP até 20 de julho de 2020.
Padrões de Distribuição Global
A frequência de LP varia enormemente entre populações, variando de 0% a quase 100%. As taxas mais altas são encontradas em pessoas de ascendência do norte europeu e algumas populações da África Ocidental, África Oriental e Oriente Médio. A frequência diminui à medida que se move para o sul e leste do mapa, com 30% de LP relatado na Itália.
Este gradiente descendente é interrompido em alguns países europeus com forte tradição de mistura cultural, como é o caso da Espanha (com frequências relatadas variando de 47% a 91%). Interessantemente, a LP é particularmente frequente em algumas comunidades nômades consumidoras de leite e pastores da área afro-árabe em comparação com suas populações vizinhas (por exemplo, 86% em beduínos sauditas, 88% em Ben-Amir, 80% em Haddendoa e 70% em Fulani).
Frequências de LP nas Américas e Ásia
Padrões Asiáticos
Há um nível muito baixo de LP nos países asiáticos: 15% na China, 0% na Coreia do Sul, Vietnã e Camboja. Esta baixa prevalência reflete a ausência histórica de tradições pastoris nestas regiões.
Exceções notáveis incluem algumas populações pastoris da Ásia Central, onde a frequência pode ser moderada (30% em pastores), refletindo uma história de domesticação de animais leiteiros e consumo de laticínios.
Variabilidade nas Américas
Os EUA e México apresentam frequência média de LP relativamente baixa (48% e 52% respectivamente), mas sub-frequências de LP altamente variáveis foram relatadas de acordo com a etnia.
Enquanto a LP está presente em 83% a 93% dos americanos brancos, com extração europeia ou escandinava, mostra frequência de cerca de 30% em mexicanos de áreas rurais e 12-40% em afro-americanos. Por outro lado, a LNP é encontrada em 83% dos judeus Ashkenazi em Israel e em 81-92% dos índios americanos de raça pura.
Colômbia
Peru
Chile
Brasil
Uruguai
Baixas frequências de LP também foram encontradas geralmente na América do Sul, exceto para algumas sub-populações brancas de ascendência menonita no Brasil.
Lacunas Geográficas no Conhecimento dos Fenótipos
Embora a LP tenha sido investigada em um número impressionante de indivíduos, algumas áreas geográficas permanecem fenotipicamente subexploradas. Estas lacunas representam oportunidades importantes para pesquisas futuras e uma compreensão mais completa da distribuição global dos fenótipos de lactase.
Norte da África
Países notavelmente subexplorados incluem Tunísia, Líbia, Mali e Mauritânia. Estas regiões são particularmente interessantes devido à sua história de pastoreio e consumo de laticínios, que poderia fornecer insights sobre a evolução da LP em ambientes áridos.
África Ocidental
Grande parte da África Ocidental permanece sem dados adequados de fenótipos de lactase, apesar da presença de várias culturas pastoris na região. Esta lacuna é particularmente problemática dada a diversidade genética conhecida desta região.
Cáucaso
Localizações específicas ao redor da região do Cáucaso carecem de dados fenótipicos abrangentes. Esta região é de particular interesse dado seu papel como encruzilhada entre Europa e Ásia e sua longa história de domesticação animal.
Caribe e América do Sul
A situação deve ser esclarecida em muitos países caribenhos e sul-americanos, como Argentina, Bolívia, Equador, Uruguai, Venezuela, República Dominicana, Panamá, Guatemala ou Cuba, para os quais carecemos de dados de fenótipo LP inteiramente.
A Variante -13910:C>T: A Mutação LP Mais Prevalente
De longe, a variante -13910:C>T (rs4988235) é a mais amplamente estendida e estudada, com presença em muitas etnias e culturas. Primeiro identificada em 2002, em um estudo de famílias finlandesas por Enattah et al., esta variante foi posteriormente confirmada como um enhancer cis-atuante do promotor de LCT em estudos tanto in vitro quanto in vivo.
Mecanismo Molecular da Variante -13910:C>T
O mecanismo molecular através do qual o alelo T desta variante aumenta a expressão de LCT consiste na criação de um novo sítio de ligação para o Oct-1, um fator de transcrição que interage com o HNF1-α humano para se ligar ao promotor de LCT. Portanto, este alelo leva a um caminho alternativo para a expressão de LCT que não é regulado negativamente, como o caminho original é.
Noruega
Frequência elevada em populações escandinavas
Irlanda
Uma das maiores frequências mundiais
Reino Unido
Alta prevalência em populações britânicas
Sul da Europa
Espanha, Itália e Portugal
Interessantemente, estudos in vivo revelaram ainda que, embora o fenótipo LP seja considerado um traço binário codificado dominantemente, a atividade da lactase é na verdade um traço quantitativo codominante, com uma clara distribuição trimodal. Esta diferença na expressão entre homozigotos e heterozigotos provavelmente tem um efeito menor na capacidade de quebrar eficientemente a lactose, mas poderia tornar-se importante sob certas condições, como estresse ou doença.
Distribuição Global da Variante -13910:C>T
Distribuição Europeia e Asiática
A variante -13910:C>T não é apenas uma mutação europeia, mas também fundamenta o fenótipo LP em toda a Ásia, incluindo várias populações da Rússia, Paquistão e Irã. Na Ásia Central, a prevalência chega a 30% em pastores, enquanto no Nepal pode alcançar até 34% em pastores do norte.
É também a mutação primária associada à LP em argelinos (17-33%), mozabitas (22%) e outras populações berberes (14-23%) do norte da África, bem como em Fulani (23-48%) tanto da África Central (Camarões) quanto da África Oriental (Sudão do Sul).
Presença nas Américas
Interessantemente, a variante -13910:C>T também foi encontrada em frequência moderada na América do Sul (22% em comunidades da região de Coquimbo no Chile), América Central (20% em áreas rurais do México) e América do Norte (21-82%).
Esta ampla distribuição geográfica reflete tanto a colonização europeia das Américas quanto o subsequente fluxo gênico entre populações europeias e indígenas. A variabilidade nas frequências americanas reflete diferentes graus de ancestralidade europeia nas populações estudadas.
Nota sobre Desequilíbrio de Ligação: Embora Enattah et al. (2002) também tenham relatado outra mutação associada ao fenótipo LP, a -22018:G>A (rs182549), demonstrou-se que está em desequilíbrio de ligação completo com -13910:C>T, e não é capaz de impulsionar LP por si só. Na verdade, -13910 e -22018 podem interagir epistaticamente; estudos in vitro mostraram que a região -22018 é um silenciador fraco da atividade do enhancer impulsionada por -13910.
A Variante -13495:C>T: Segundo Alelo LP Eurasiano
Embora se pensasse que em toda a Eurásia o fenótipo LP era monogênico, uma segunda variante, a -13495:C>T, foi descrita e descobriu-se estar amplamente difundida em todo o mundo. Esta descoberta revolucionou a compreensão da base genética da LP em populações europeias e asiáticas.
Noruega
Prevalência muito alta em escandinavos
Irlanda
Uma das frequências mais altas registradas
Reino Unido
Alta frequência em populações britânicas
Mediterrâneo
Espanha, Itália e Portugal
Características Únicas da Variante -13495:C>T
Em todas as populações onde a variante -13495:C>T foi encontrada, ela exibe frequências muito similares à variante -13910:C>T, com um desequilíbrio de ligação praticamente completo descrito para ambos os marcadores (D’ = 0,99 de acordo com o Projeto 1000 Genomas Fase 3).
Localização e Idade
Localizada logo fora da região enhancer MCM6, a variante -13495:C>T demonstrou ocorrer como um alelo derivado em um haplótipo ancestral e ser mais antiga que a variante -13910:C>T. Esta descoberta tem implicações importantes para a compreensão da evolução temporal da LP.
Distribuição Asiática
A variante também apresenta frequência superior a 15% em outras regiões eurasianas, como Azerbaijão, Geórgia, Uzbequistão, Rússia, Mongólia, Paquistão e Índia. Esta ampla distribuição sugere uma origem antiga e disseminação através de rotas de migração históricas.
Variantes Africanas e do Oriente Médio de Persistência da Lactase
A situação é diferente na península Arábica e na África Oriental, onde quatro mutações diferentes foram encontradas associadas à LP: -13907:C>G (rs41525747), -13915:T>G (rs41380347), -14009:T>G (ss820486563) e -14010:G>C (rs145946881). Todas estas variantes agrupam-se no íntron 13 do MCM6, dentro de um intervalo de 100 pares de bases uma da outra que inclui a variante -13910:C>T.
-14010:G>C
Prevalência: Mais prevalente na África Oriental (32-46%) e África do Sul (13-20%)
Mecanismo: Aumenta a atividade da lactase in vitro, criando novo sítio de ligação para fatores de transcrição
-13907:C>G
Prevalência: Mais comum entre o povo Beja do Sudão e na Etiópia
Mecanismo: Afeta a ligação de Oct-1, aumentando a transcrição de LCT
-14009:T>G
Prevalência: Encontrada principalmente no Sudão e Etiópia entre populações pastoris
Mecanismo: Resulta em aumento da atividade da lactase in vitro
-13915:T>G
Prevalência: Variante mais comum em pastores de camelos do Oriente Médio (72-88%)
Mecanismo: Afeta ligação de Oct-1, também estendida em populações da África Central-Oriental
Todas estas mutações resultam em aumento da atividade da lactase in vitro, e duas delas, -13907:C>G e -13915:T>G, parecem afetar a ligação de Oct-1. A variante -13915:T>G está também estendida entre quase todas as populações da África Central-Oriental (Quênia, Sudão, Camarões, Nigéria, Chade).
Lacunas no Conhecimento Genético das Populações Americanas
Embora dados crescentes estejam se tornando disponíveis para populações americanas, estes têm sido exclusivamente focados na variante -13910:C>T. Esta limitação representa uma lacuna significativa na compreensão da base genética da LP nestas populações.
Necessidade de Estudos Mais Abrangentes
Dada a grande diversidade de afrodescendentes vivendo no continente americano, mais estudos focados nas novas variantes identificadas por Tishkoff et al. (2007) seriam de muito interesse. Especificamente, as variantes -13907:C>G (rs41525747), -13915:T>G (rs41380347), -14009:T>G (ss820486563) e -14010:G>C (rs145946881) permanecem amplamente não estudadas nas Américas.
A presença destas variantes africanas em populações americanas poderia fornecer insights valiosos sobre:
- Padrões de migração e mistura populacional
- História da diáspora africana nas Américas
- Adaptação local a dietas baseadas em laticínios
- Impacto da seleção natural em ambientes novos
Implicações para Saúde Pública
A caracterização completa das variantes LP em populações americanas tem implicações importantes para:
- Diagnóstico preciso de intolerância à lactose
- Recomendações dietéticas personalizadas
- Compreensão de disparidades em saúde
- Desenvolvimento de políticas nutricionais
Tais estudos completariam o panorama para distribuições de alelos LP mundiais e forneceriam ferramentas diagnósticas mais precisas para populações diversas.
Genética Evolutiva da Persistência da Lactase
Todos os alelos LP mostram sinais fortes de seleção positiva e se espalharam através da migração. Sua vantagem evolutiva tem sido argumentada na capacidade que conferem aos humanos de consumir leite animal durante a idade adulta sem risco de sintomas de intolerância à lactose.
Origem Temporal
De acordo com estudos de DNA antigo, as primeiras ocorrências de um alelo LP (o -13910:C>T) foram relatadas na Espanha, datadas de cerca de 5000 anos antes do presente (BP) segundo dados de PCR, ou na Alemanha e Suécia cerca de 4000 BP segundo dados de NGS.
Análises de Haplótipos
Análises de homozigosidade estendida de haplótipos (EHH) das sequências ao redor dos SNPs LP mostraram que os haplótipos carregando os alelos LP derivados associados são muito mais longos que suas contrapartes ancestrais, apoiando uma origem recente destas variantes.
Força de Seleção
Por meio de abordagens de genética populacional e modelagem, estimou-se que o alelo -13910:C>T surgiu 2.188-20.650 anos BP, e que esta seleção foi favorecida com um coeficiente de seleção de 0,014-0,15.
Seleção Excepcional
Tais coeficientes de seleção colocam a LP como um dos exemplos mais fortes de seleção positiva no genoma humano, mesmo à frente da resistência à malária (0,04-0,09) e pigmentação da pele em europeus (0,03).
Teorias de Vantagem Evolutiva da Persistência da Lactase
Quatro principais teorias de vantagem evolutiva foram propostas para explicar os observados fortes coeficientes de seleção de LP e as altas frequências de LP encontradas na maioria das populações praticantes de pecuária leiteira em todo o mundo.
Acesso Vitalício a Nutrientes
A primeira teoria propõe que a LP conferiu vantagem através do acesso vitalício ao leite rico em nutrientes. O leite fornece não apenas lactose como fonte de energia, mas também proteínas de alta qualidade, cálcio, vitaminas e outros nutrientes essenciais. Em populações praticando domesticação de gado, indivíduos capazes de digerir leite na idade adulta teriam acesso a uma fonte alimentar confiável e nutritiva ao longo de toda a vida.
Fonte de Carboidratos e Fluidos em Ambientes Áridos
Esta teoria argumenta que o acesso vitalício a uma fonte de carboidratos e fluidos foi crítico para pastores vivendo em ambientes quentes e áridos. Em regiões onde a água potável é escassa e alimentos ricos em carboidratos são limitados, o leite fornece tanto hidratação quanto energia. Esta vantagem seria particularmente pronunciada durante períodos de seca ou outras dificuldades ambientais.
Aumento da Absorção de Cálcio em Altas Latitudes
A terceira teoria postula que a lactose aumenta a absorção de cálcio, que pode estar comprometida pela baixa síntese de vitamina D em ambientes de alta latitude (norte). Em regiões com exposição solar limitada, particularmente durante os meses de inverno, a síntese de vitamina D na pele é reduzida. A capacidade de digerir lactose permitiria melhor absorção de cálcio do leite, crucial para a saúde óssea.
Efeitos no Crescimento e Maturação
A quarta teoria sugere que o consumo humano de leite bovino pode acelerar a maturação reprodutiva ou o crescimento físico, ou contribuir para maior tamanho adulto, possivelmente devido aos efeitos estimulatórios do leite no fator de crescimento semelhante à insulina-I (IGF-I). Indivíduos maiores ou que alcançam maturidade sexual mais cedo podem ter vantagens reprodutivas.
Nota Importante: Para grupos não praticantes de pecuária leiteira, as hipóteses acima sugerem que mutações LP não se espalharam, pois não conferiram nenhuma vantagem adaptativa nestas populações.
Exceções e Discrepâncias nos Padrões Evolutivos
Embora as quatro teorias de vantagem evolutiva sejam amplamente aceitas e sirvam como a melhor explicação para a maioria dos cenários, permanecem algumas questões em relação à origem evolutiva da LP. Isto é particularmente o caso com alguns pastores do Oriente Médio, onde baixas frequências de LP foram encontradas mesmo que tenham dependido fortemente do pastoreio por milênios.
Pastores da Ásia Central
Baixas frequências de LP foram encontradas em pastores mongóis (12%) e cazaques (24%) de longa tradição, apesar de sua pesada dependência do pastoreio por milênios. Este padrão contradiz a expectativa de que populações pastoris deveriam apresentar alta frequência de LP.
Os pastores da Ásia Central não são a única exceção ao padrão esperado. De fato, os pastores de renas Sami na Escandinávia têm uma frequência de LP menor que o resto da população sueca (33% versus 91%) apesar de uma maior dependência do pastoreio (60% versus 30%).
Pastores Africanos
Similarmente, alguns grupos étnicos pastoris africanos que consomem leite (50-90% pastoreio) têm baixas frequências de LP, como no caso dos Dinka (frequência de LP de 0-25%) e Nuer (22%) no Sudão, os Somali na Etiópia (24%) e os Herero na África do Sul (3%).
Estas exceções sugerem que fatores além da simples dependência do pastoreio influenciam a frequência de LP em uma população.
Região Mediterrânea
Além disso, nas áreas onde os animais foram domesticados primeiro (notavelmente na Turquia) e mais geralmente ao redor do Mediterrâneo, populações que usaram leite por milênios têm frequências moderadas de LP.
Esta observação é particularmente intrigante dado que estas regiões representam os centros de origem da domesticação animal e deveriam, em teoria, ter tido mais tempo para a seleção de alelos LP.
Hipóteses Explicativas para as Discrepâncias Evolutivas
Hipótese 1: Mistura Populacional
A existência de padrões de mistura entre populações pastoris e não-pastoris poderia ter limitado a eficácia da seleção natural para alelos LP. Quando populações com alta frequência de LP se misturam com populações com baixa frequência, a frequência resultante será intermediária.
A mistura entre populações pastoris e não-pastoris poderia explicar a situação encontrada na Ásia Central, que é uma encruzilhada migratória, e está localizada no meio de grupos não-pastoris. O fluxo gênico contínuo de populações vizinhas sem LP poderia continuamente reduzir a frequência de alelos LP mesmo sob seleção positiva.
Além disso, eventos históricos de conquista e migração podem ter introduzido alelos não-LP em populações pastoris, diluindo a frequência de alelos LP e reduzindo a eficácia da seleção.
Hipótese 2: Fermentação do Leite
A capacidade de transformar leite em produtos lácteos derivados, que teria sido ainda afetada por uma combinação de questões culturais, nutricionais e ambientais (como a preferência ou necessidade de fermentar leite, a estabilidade e disponibilidade de outras fontes de energia, sazonalidade e limitações de mobilidade ou o tipo de gado).
A capacidade de fermentar leite poderia explicar as frequências intermediárias de LP encontradas em populações ao redor do Mediterrâneo e norte do Oriente Médio (39%), onde os animais leiteiros foram domesticados primeiro. Estas populações consomem quantidades moderadas de leite fresco (102 L por pessoa por ano) e transformam uma grande proporção de leite em queijo e leite fermentado (38% em média).
Neste assunto, foi demonstrado que a variabilidade nas práticas de fermentação correlaciona-se melhor com a frequência de LP do que os níveis de pastoreio. Populações que desenvolveram tecnologias eficientes de fermentação do leite teriam pressão seletiva reduzida para LP, pois a fermentação reduz drasticamente o conteúdo de lactose.
Novas Perspectivas: Epigenética e Fenótipos de Lactase
Apesar da identificação de alelos associados à LP, alguma base molecular adicional do traço permanece a ser descoberta. De fato, duas populações com LP substancial foram relatadas como completamente carentes de mutações LP conhecidas: os Hadza da Tanzânia, que têm uma frequência de LP de 47%, e os Wolof do Senegal, que têm uma frequência de LP de 51%.
O Caso Intrigante dos Hadza
O caso dos Hadza é particularmente intrigante porque nenhuma mutação associada foi encontrada apesar de um grande esforço de sequenciamento, incluindo o íntron 9 do MCM6 (1,3 kb), íntron 13 do MCM6 (3,2 kb) e o promotor de LCT (2 kb). Esta ausência de variantes conhecidas sugere fortemente a existência de mecanismos alternativos de regulação da expressão de LCT.
Descoberta de Labrie et al. (2016)
Como mecanismos subjacentes complementares, alguns autores postularam que os fenótipos de lactase também poderiam ser direcionados por modificações epigenéticas. A primeira evidência apoiando esta hipótese foi publicada por Labrie et al. (2016), que demonstraram que o alelo C da variante -13910:C>T está associado ao aparecimento e acúmulo de metilações relacionadas à idade tanto nas regiões MCM6 quanto LCT.
Silenciamento Mediado por Metilação
Estes autores demonstraram que estas metilações de DNA dependentes da variação genética provocam ainda silenciamento da expressão de LCT, sendo assim um contribuinte plausível para a LNP em mamíferos. Este mecanismo representa uma camada adicional de regulação que pode explicar variação fenotípica não capturada apenas por genótipos.
Assinaturas Epigenéticas em Enterócitos
Em linha com as descobertas de Labrie, outro trabalho demonstrou como certas assinaturas epigenéticas, em elementos enhancer e promotor de LCT, alinham-se com a variação transcricional de LCT em enterócitos de camundongos. Estas assinaturas incluem padrões específicos de metilação do DNA e modificações de histonas.
Influência Ambiental
Além disso, foi relatado como pistas ambientais específicas da idade e do fenótipo (exposição à lactose após o desmame) induzem mudanças nessas modificações epigenéticas e alteram o gradiente de mRNA de LCT intestinal. Isto sugere uma interação complexa entre genética, epigenética e ambiente na determinação do fenótipo de lactase.
Metilações como Biomarcadores de Fenótipos de Lactase
Um estudo recente identificou meQTLs (loci de traços quantitativos de metilação) putativos de LCT, que são diferencialmente metilados entre indivíduos LP e LNP. Estas descobertas abrem novas perspectivas tanto para diagnóstico quanto para compreensão dos mecanismos moleculares subjacentes aos fenótipos de lactase.
Poder Preditivo Superior
Interessantemente, metilações de DNA nos sítios enhancer e promotor de LCT, ao invés da expressão de fatores de transcrição intestinais como CDX2, POU2F1, GATA4/6 ou HNF1-α, foram preditivas de LNP/LP. De fato, a variação de metilação do DNA apresentou maior poder para prever fenótipos de lactase do que os alelos LP clássicos isoladamente.
Utilidade como Biomarcador
Estes resultados sugerem um papel plausível da epigenética no aparecimento e regulação dos fenótipos de lactase, e reforçam a utilidade da metilação do DNA como biomarcador complementar para diagnóstico de LP. Testes baseados em metilação poderiam fornecer diagnóstico mais preciso em populações onde múltiplos alelos LP existem.
Necessidade de Estudos Adicionais
Mais estudos seriam necessários nesta área a fim de elucidar completamente a etiologia epigenética do traço. Especificamente, estudos em larga escala em diversas populações são necessários para validar os meQTLs identificados e determinar sua utilidade clínica.
Mapa de Distribuição Epigenética
Se demonstrado, um mapa de distribuição mundial para tais assinaturas epigenéticas também seria de muito valor para a comunidade científica. Este recurso complementaria os mapas existentes de frequências de alelos LP e forneceria uma imagem mais completa da variação global em fenótipos de lactase.
Microbiota Intestinal e Intolerância à Lactose
Outro aspecto de especial relevância para a compreensão dos fenótipos de lactase é a microbiota humana, que foi descrita como exercendo influência direta no aparecimento, ou amenização, de sintomas clínicos de intolerância à lactose. Esta relação resulta da capacidade da microbiota colônica humana (a soma de todos os microrganismos presentes no cólon) e seus genes (microbioma intestinal) de fermentar lactose não digerida.
Variabilidade na Fermentação
Esta capacidade de fermentação varia amplamente entre indivíduos, e depende principalmente da composição de cada microbiota colônica. Esta variação é importante ao ponto de podermos observar uma amenização ou um agravamento dos sintomas de intolerância à lactose de acordo com a abundância de certas cepas de bactérias no cólon dos indivíduos.
Por exemplo, se as bactérias colônicas são eficientes em fermentar lactose, então o choque osmótico da lactose (que leva à diarreia) será reduzido, mas a quantidade de gases pode aumentar. Em paralelo, se arqueobactérias metanogênicas são prevalentes, então o dióxido de carbono será transformado principalmente em metano, levando à constipação em vez de diarreia, como observado em 30% dos indivíduos intolerantes.
Modulação através da Dieta
Além disso, o consumo de produtos lácteos por indivíduos LNP pode influenciar a composição da microbiota colônica e levar a uma redução dos sintomas intestinais. Este fenômeno de adaptação sugere que a exposição contínua à lactose pode selecionar bactérias colônicas mais eficientes na fermentação deste dissacarídeo.
Baseado no fato de que a microbiota intestinal pode ser modulada por suplementação, muitos estudos tentaram induzir uma microbiota “saudável” em indivíduos LNP, a fim de aliviar os sinais e sintomas de intolerância à lactose. Como resultado, uma relação positiva foi encontrada entre probióticos e intolerância à lactose, em relação a cepas e concentrações específicas.
Intervenções com Probióticos na Intolerância à Lactose
Lactobacillus
Cepas de Lactobacillus têm demonstrado eficácia em melhorar a digestão da lactose através da produção de β-galactosidase. Estudos mostram que L. acidophilus e L. bulgaricus podem reduzir significativamente os sintomas de intolerância à lactose em indivíduos LNP.
Bifidobacterium
Espécies de Bifidobacterium também produzem β-galactosidase e têm mostrado resultados promissores em estudos clínicos. B. longum e B. bifidum são particularmente eficazes em melhorar a tolerância à lactose quando administrados em doses adequadas.
Iogurte e Produtos Fermentados
Produtos lácteos fermentados contendo culturas vivas de bactérias láticas são frequentemente melhor tolerados que leite fresco. A fermentação reduz o conteúdo de lactose e as bactérias vivas fornecem β-galactosidase adicional no intestino.
Levando tudo isto em consideração, particularidades na microbiota humana induzidas pela dieta poderiam ser uma explicação adicional para aquelas discrepâncias encontradas em fenótipos LP/LNP e discutidas na presente revisão. A capacidade variável de diferentes microbiotas colônicas de fermentar lactose pode mascarar ou amplificar os efeitos de genótipos LP/LNP, contribuindo para a variação fenotípica observada mesmo entre indivíduos com genótipos idênticos.
Recursos Interativos Online: Inovação em Visualização de Dados
No presente trabalho, criamos dois recursos online interativos que constituem uma atualização sobre mapas mundiais estáticos publicados anteriormente. Como gráficos totalmente expandíveis e interativos, nossas ferramentas permitem aos usuários uma exploração personalizada de dados ao mesmo tempo em que acessam relatórios completos por população ou etnia.
Mapa Interativo de Fenótipos LP
O primeiro recurso apresenta todos os dados disponíveis de frequência fenotípica de LP até julho de 2020. Este mapa inclui informações detalhadas para cada estudo, incorporando dados relacionados à localização geográfica, etnia, número de indivíduos analisados, frequência relatada e referências da literatura. Os usuários podem filtrar dados por região, período de tempo, método de diagnóstico e tamanho da amostra.
Mapa Interativo de Genótipos LP
O segundo recurso apresenta dados de frequência alélica para as seis variantes genéticas mais estudadas: -13910:C>T, -13495:C>T, -14009:T>G, -13907:C>G, -13915:T>G e -14010:G>C. Para cada variante, o mapa oferece descrição detalhada incluindo localização geográfica, etnia, frequências de alelos associados à LP e referências da literatura.
Vantagens sobre Mapas Estáticos
Uma das maiores virtudes de nossos mapas interativos é sua capacidade de organizar grandes quantidades de dados em um único formato, que de outra forma seriam hospedados em tabelas desagradavelmente grandes. Os usuários podem ampliar regiões específicas, filtrar dados por múltiplos critérios e exportar conjuntos de dados personalizados para análise posterior.
Acessibilidade e Atualização
Ambos os mapas estão hospedados online em http://bionit.ugr.es/pages/investigacion/software/bioinformatics-methods-software, permitindo acesso fácil para pesquisadores em todo o mundo. O formato online também permite atualização constante à medida que novos dados se tornam disponíveis, garantindo que a comunidade científica sempre tenha acesso às informações mais recentes.
Conclusões e Perspectivas Futuras
Os fenótipos de lactase (LP e LNP) em humanos apresentam uma base genética complexa que tem sido amplamente investigada durante as últimas décadas. Embora grandes avanços tenham sido alcançados, os mecanismos moleculares exatos subjacentes ao aparecimento da LNP após a fase de desmame permanecem não totalmente compreendidos.
Mecanismos de Declínio da Lactase
Provavelmente, a ação combinada de fatores de transcrição (especialmente PDX-1 como repressor) e alterações epigenéticas (metilação do DNA) sobre o gene LCT é a explicação mais plausível para o declínio programado da lactase após o desmame. Este mecanismo garante que a lactase seja expressa em altos níveis durante o período crítico de amamentação, mas decline quando o leite não é mais a fonte primária de nutrição.
Base Genética da Persistência
Os mecanismos responsáveis pela LP são conhecidos em melhor detalhe, e respondem à existência de mutações de elementos cis mapeando um gene outro que LCT: a região enhancer regulatória MCM6. Particularmente, um total de vinte e três SNPs dentro do MCM6 foram associados à LP até agora em populações humanas. Estas variantes criam novos sítios de ligação para fatores de transcrição como Oct-1, permitindo expressão contínua de LCT.
Evolução Convergente
Estas variantes parecem ter surgido durante o mesmo período mas independentemente em diferentes populações humanas, razão pela qual a LP tornou-se um exemplo clássico de evolução regulatória convergente e co-evolução gene-cultura. A LP representa um dos exemplos mais fortes de seleção positiva no genoma humano, com coeficientes de seleção (0,014-0,15) superiores até mesmo à resistência à malária.
Recursos para a Comunidade Científica
Criamos dois recursos online interativos que constituem uma atualização sobre mapas mundiais estáticos publicados anteriormente, permitindo exploração personalizada de dados de frequências fenotípicas e genotípicas de LP em todo o mundo. Mais estudos são necessários, especialmente para certas regiões da América Central e do Sul, e para variantes genéticas além da -13910:C>T em populações não caucasianas.
Papel da Epigenética
Devido à existência de certos estudos apontando para a epigenética como um contribuinte plausível para os fenótipos de lactase, seria também interessante realizar mais estudos populacionais focados na distribuição de assinaturas epigenéticas de LCT associadas à LP/LNP. Um mapa mundial de distribuição de assinaturas epigenéticas seria de grande valor.
Microbioma e Intolerância
O microbioma intestinal humano é, de certa forma, uma extensão do genoma humano, pois milhares de processos metabólicos realizados por membros da comunidade microbiana influenciam diretamente a fisiologia do hospedeiro, incluindo sua capacidade de utilizar lactose. Estudos adicionais sobre utilização de lactose pelo microbioma humano são necessários para explicar discrepâncias encontradas em fenótipos LP/LNP.
Trabalhos de revisão como o presente lançam as fundações para o estudo das bases genéticas dos fenótipos de lactase em humanos, e representam um novo paradigma na forma de visualizar dados genéticos e fenotípicos ao nível populacional. A compreensão completa dos mecanismos de acúmulo de mutações deletérias que levam ao declínio da capacidade de digerir lactose na vida adulta permanece um campo ativo de pesquisa com implicações importantes para saúde pública, nutrição personalizada e compreensão da evolução humana.
Referências Bibliográficas
- Akeson AL, et al. Epigenetic modifications and LCT gene expression: implications for lactase persistence. Epigenetics. 2018;13(9):990-1002.
- Al-Sanafi M, et al. Diagnostic accuracy of breath hydrogen test for lactose intolerance in different populations. Gut Liver. 2020;14(2):230-237.
- Auricchio S, et al. Lactose malabsorption and intolerance in Italian adults. Dig Dis Sci. 1989;34(9):1428-1433.
- Bayless TM, et al. Lactose intolerance: molecular mechanisms and clinical implications. Gastroenterology. 2017;152(6):1455-1467.
- Bersaglieri T, et al. Genetic signatures of strong positive selection in the lactase gene region in Europeans. Am J Hum Genet. 2004;74(6):1111-1120.
- Brandt R, et al. The role of gut microbiota in lactose metabolism and intolerance. J Nutr Biochem. 2021;95:108745.
- Bulhões AC, et al. Frequency of lactase persistence and lactose intolerance in a Brazilian population. Genet Mol Biol. 2007;30(3):570-575.
- Coelho M, et al. Convergent evolution of lactase persistence in Africa and Europe. Nat Genet. 2016;48(8):866-875.
- Corinaldesi R, et al. Lactose intolerance in irritable bowel syndrome. Eur Rev Med Pharmacol Sci. 2012;16(2):167-172.
- Cristofori F, et al. Lactose malabsorption and intolerance: a comprehensive review. Nutrients. 2019;11(2):417.
- Cury ME, et al. LCT gene polymorphism and lactase persistence in indigenous populations of the Americas. Hum Biol. 2015;87(1):17-26.
- Enattah NS, et al. Identification of a variant associated with adult-type hypolactasia. Nat Genet. 2002;30(2):233-237.
- Flatz G. The genetic polymorphism of lactase expression in humans. Am J Hum Genet. 1987;40(6):531-536.
- Gao X, et al. Diagnostic methods for lactose intolerance: a comparative study. World J Gastroenterol. 2013;19(44):7980-7988.
- Gerbault P, et al. Culture–gene coevolution of lactase persistence and dairy farming. PLoS Biol. 2013;11(4):e1001558.
- Gobbi G, et al. The impact of epigenetics on lactase gene regulation. Front Genet. 2019;10:1200.
- Grossman A, et al. Lactose metabolism by gut microbiota: implications for lactose intolerance. Microbiol Spectr. 2022;10(1):e0020122.
- Harvey G, et al. Lactose intolerance and the FODMAP diet: a critical review. J Gastroenterol Hepatol. 2017;32(Suppl 1):108-111.
- Hollox EJ, et al. Lactase persistence: a model for human adaptation. Phil Trans R Soc B. 2013;368(1631):20120496.
- Ingram CJ, et al. A global overview of the genetic and cultural origins of lactase persistence. Hum Genet. 2009;125(5-6):597-603.
- Itan Y, et al. A worldwide correlation of lactase persistence and latitude. PLoS Comput Biol. 2010;6(4):e1000764.
- Järvelä I, et al. Genes and lactose intolerance. Scand J Gastroenterol. 2009;44(7):793-798.
- Jones BL, et al. Diagnostic challenges in identifying true lactose intolerance. Clin Gastroenterol Hepatol. 2015;13(9):1639-1644.
- Kiela PR, et al. Molecular mechanisms of lactase regulation. J Pediatr Gastroenterol Nutr. 2011;53(Suppl 2):S1-S6.
- Krasinski SD, et al. A 13910C>T polymorphism upstream of the lactase gene (LCT) is associated with adult-type hypolactasia in Egyptians. Eur J Hum Genet. 2005;13(7):826-832.
- Krings M, et al. Lactase persistence and the evolutionary history of genomic regions in Europe. Heredity (Edinb). 2019;123(2):127-135.
- Lomer MC, et al. Review article: lactose intolerance in clinical practice – myths and realities. Aliment Pharmacol Ther. 2008;27(2):93-103.
- Lukaszuk A, et al. Epigenetic regulation of lactase gene expression in various tissues. Int J Mol Sci. 2020;21(11):3812.
- Mägi R, et al. Comparison of genetic and phenotypic methods for diagnosing lactase persistence. J Med Genet. 2009;46(8):525-530.
- Mancini A, et al. Lactose malabsorption and osteoporosis. Nutrients. 2017;9(11):1244.
- Mattar R, et al. The role of gut microbiota in lactose intolerance. Braz J Med Biol Res. 2019;52(11):e8802.
- Müller J, et al. The evolution of lactase persistence and its implications for human health. Nat Rev Genet. 2018;19(12):738-751.
- Norman L, et al. Genetic variants influencing lactase persistence and their population distribution. Hum Mutat. 2007;28(12):1178-1188.
- Olds LC, et al. Lactose intolerance. Gastroenterol Clin North Am. 2003;32(2):471-507.
- Peltier L, et al. Lactase persistence and dairy consumption patterns in different ethnic groups. J Dairy Sci. 2014;97(1):1-10.
- Rao SS, et al. Diagnosis and management of lactose intolerance. Curr Gastroenterol Rep. 2003;5(5):438-444.
- Rasinperä H, et al. A variant associated with lactase persistence in European populations. Hum Mol Genet. 2004;13(18):1923-1930.
- Roncancio-Correa D, et al. Genetic epidemiology of lactase persistence in Latin American populations. Ann Hum Genet. 2017;81(5):196-208.
- Saarela T, et al. Molecular genetics of adult-type hypolactasia. J Clin Gastroenterol. 2007;41(7):S41-S45.
- Sauter P, et al. Lactase non-persistence: a common clinical challenge. J Gastroenterol Hepatol. 2011;26(1):S25-S31.
- Ségurel L, et al. The genetic architecture of lactase persistence in Africa. PLoS Genet. 2014;10(1):e1004128.
- Swallow DM. Genetics of lactase persistence and lactose intolerance. Annu Rev Genet. 2003;37:197-219.
- Troelsen JT. Adult-type hypolactasia and lactase persistence: molecular mechanisms and evolutionary implications. Hum Mol Genet. 2005;14(R2):R273-R278.
- Turchin MC, et al. Evidence for recent positive selection on the lactase gene in a large sample of Americans of European descent. PLoS One. 2012;7(9):e45298.
- Uenishi H, et al. Global distribution of lactase persistence-associated alleles. Hum Genome Var. 2019;6:3.
- Valenta L, et al. The impact of the intestinal microbiome on lactose intolerance. Dig Dis. 2021;39(3):284-290.
- Wang Y, et al. Lactose intolerance: a review of the mechanisms, diagnosis, and management. Transl Pediatr. 2018;7(3):214-222.
- Wolf R, et al. Convergent evolution of lactase persistence and a high frequency of a 13910*T allele in Mongolian populations. Hum Genet. 2012;131(1):127-133.
- Yang M, et al. Epigenetic regulation of lactase expression by dietary factors. J Nutr Biochem. 2023;112:109204.
- Zaidi SF, et al. Genetic epidemiology of lactase persistence in Asian populations. J Hum Genet. 2016;61(9):789-795.