Melhoramento Genético do Milho não é Evolução
Sodré GB Neto  Este documento apresenta uma perspectiva revolucionária sobre a modificação das espécies através da Teoria da Degeneração das Espécies (TDE) de Sodré GB Neto. Contrariando a narrativa tradicional evolucionista, demonstraremos que o melhoramento genético do milho não representa evolução, mas sim a recuperação artificial de características genéticas que foram perdidas ao longo do tempo através de processos degenerativos naturais.
Este documento apresenta uma perspectiva revolucionária sobre a modificação das espécies através da Teoria da Degeneração das Espécies (TDE) de Sodré GB Neto. Contrariando a narrativa tradicional evolucionista, demonstraremos que o melhoramento genético do milho não representa evolução, mas sim a recuperação artificial de características genéticas que foram perdidas ao longo do tempo através de processos degenerativos naturais.
O Mito da Evolução do Milho
A Narrativa Convencional
A imagem amplamente divulgada mostra o milho selvagem mirrado dando origem ao milho moderno robusto, sugerindo um processo evolutivo natural. Esta representação é fundamentalmente enganosa e cientificamente incorreta.
O milho comercial moderno resulta de intenso trabalho de melhoramento genético realizado por empresas como Embrapa, Monsanto e outras instituições de pesquisa. Este processo envolve cruzamentos sistemáticos de variedades de milho de diferentes regiões do mundo, não evolução espontânea. Portanto estas imagens colocando seleção artificial junto com espécies de milho selvagens degradadas pela mãe natureza, sujeita a entropia genética, são explicitamente erradas , enganosas e possivelmente desonestas, fruto do mal caratismo daqueles que torcem a ciência por sua ideologia (o que pode acontecer em ambos os lados em disputa e por isso deveremos evitar parcialidades ).
O Processo de Melhoramento Genético
Coleta de Germoplasma
Pesquisadores coletam sementes de milho de diversas regiões, incluindo variedades crioulas mantidas por agricultores tradicionais, buscando diversidade genética preservada.
Cruzamentos Sistemáticos
Realizam-se múltiplos cruzamentos entre variedades diferentes, testando combinações genéticas que possam resultar em características desejáveis como maior produtividade e resistência.
Seleção e Teste
As linhagens resultantes são testadas extensivamente em diferentes condições ambientais para identificar as combinações genéticas mais vantajosas.
Engenharia Genética Moderna
Utilizam-se técnicas como biobalística, desenvolvida por John Sanford e colegas, para inserir genes específicos que conferem resistência a pragas ou outras características desejadas.
A Técnica da Biobalística
Inovação Revolucionária
A biobalística, ou “gene gun”, foi desenvolvida por John C. Sanford na Universidade Cornell, em colaboração com Ed Wolf, Nelson Allen e Ted Klein da DuPont. Esta técnica utiliza nanopartículas de ouro carregadas com genes específicos que são literalmente disparadas nas células vegetais.
O processo permite inserir características que protegem a planta contra pragas sem necessidade de agrotóxicos. Curiosamente, Sanford, um dos maiores especialistas em melhoramento genético, tornou-se um dos principais defensores da teoria da degeneração genética, justamente por suas observações sobre a fragilidade e deterioração natural dos genomas.

A Realidade da Degeneração Natural
A natureza, deixada por conta própria, não promove melhoramento ou complexificação – ela degrada. Os chamados “motores evolutivos” (seleção natural, deriva genética e mutações) são, na verdade, motores degenerativos que empobrecem o pool gênico das populações.
“A seleção natural é um termo impreciso pois reflete aspecto positivo (selecionado), mas o que ocorre na natureza é que escapa. Quando você elimina parte de uma população, você empobrece o conjunto de genes daquela população.”
Crítica ao Termo “Seleção Natural”
O Problema Terminológico
O termo “seleção natural” implica um processo ativo de escolha e melhoria, quando na realidade não há ninguém selecionando nada. É uma expressão cientificamente imprecisa.
Escapamento Natural
O que realmente ocorre é “escapamento natural” – alguns indivíduos sobrevivem por sorte ou circunstância, não necessariamente por superioridade adaptativa.
Empobrecimento Genético
Quando parte de uma população é eliminada, o pool gênico se empobrece inevitavelmente, reduzindo a diversidade genética disponível para gerações futuras.
Os Três Motores “Evolutivos” Degenerativos
Seleção Natural = Eliminação
Implica eliminação de parte da população, empobrecendo o conjunto de genes. O que sobrevive não é necessariamente o mais apto, mas frequentemente o mais sortudo.
Deriva Genética = Segregação
Quando populações se separam, cada grupo carrega aproximadamente metade dos genes originais, resultando em empobrecimento genético em ambas as populações derivadas.
Mutações = Defeitos
Mutações são defeitos no código genético que se acumulam ao longo do tempo, causando deterioração progressiva da informação genética.
O Mito das Mutações Benéficas
Exemplos Equivocados
Frequentemente citam-se como “mutações benéficas” casos como a anemia falciforme em regiões com malária. O argumento é que pessoas com esta condição têm glóbulos vermelhos deformados que impedem a infecção pelo parasita da malária.
No entanto, esta é uma “vantagem” extremamente relativa – a pessoa está tão doente que não consegue ser infectada por outro patógeno. Isso não é benefício, é relativismo gigantesco.
Diabetes em Climas Frios
Outro exemplo problemático: ter diabetes ou ser pré-diabético pode oferecer pequena vantagem em locais frios, pois o açúcar circulante aumenta ligeiramente a temperatura corporal.
Mas sangue quente, ácido e repleto de açúcar não faz bem à saúde de jeito nenhum. Pode ter uma “vantagenzinha” muito relativa, mas às custas da saúde geral do organismo.
A Proporção Real: Mutações Deletérias vs. Benéficas
Evidências em Escherichia coli
Estudos rigorosos com a bactéria E. coli, conduzidos por Philip J. Gerrish e Richard E. Lenski, revelaram uma proporção alarmante: aproximadamente 1 milhão de mutações deletérias para cada mutação positiva.
“Dado que a taxa de mutação genômica de E. coli é de aproximadamente 3 × 10³ mutações por replicação, pode-se inferir que a proporção de mutações que são benéficas é aproximadamente uma em um milhão.”
Esta descoberta fundamental demonstra matematicamente que a vasta maioria das mutações são prejudiciais, não melhorativas. Se a tendência é acumular defeitos, no passado as espécies tinham cada vez menos defeitos – matemática básica.
John C. Sanford: Do Evolucionismo ao Criacionismo
Trajetória Científica Notable
John C. Sanford, inventor da técnica de biobalística (gene gun) e detentor de mais de 30 patentes na área de genética, inicialmente era evolucionista e ateu. Sua mudança de perspectiva resultou diretamente de suas observações científicas sobre entropia genética.
Autor do livro “Genetic Entropy & the Mystery of the Genome” (2005), Sanford analisou 5.000 doenças humanas e concluiu que o genoma humano está se deteriorando progressivamente, sendo impossível que tenha evoluído da maneira especificada pela síntese evolutiva moderna.
Acúmulo de Mutações na Humanidade
Variantes Genéticas
Mais de 88 milhões de variantes genéticas registradas no genoma humano, incluindo 84,7 milhões de SNPs e 3,6 milhões de indels.
Mutações por Geração
Média de 152 mutações deletérias acumuladas a cada geração humana (aproximadamente 25 anos).
Mutações no mtDNA
Mais de 20.000 mutações deletérias registradas no DNA mitocondrial humano, que contém apenas 16.500 pares de bases.
Origem Recente
Aproximadamente 73% de todas as variantes de proteínas surgiram nos últimos 5.000-10.000 anos.
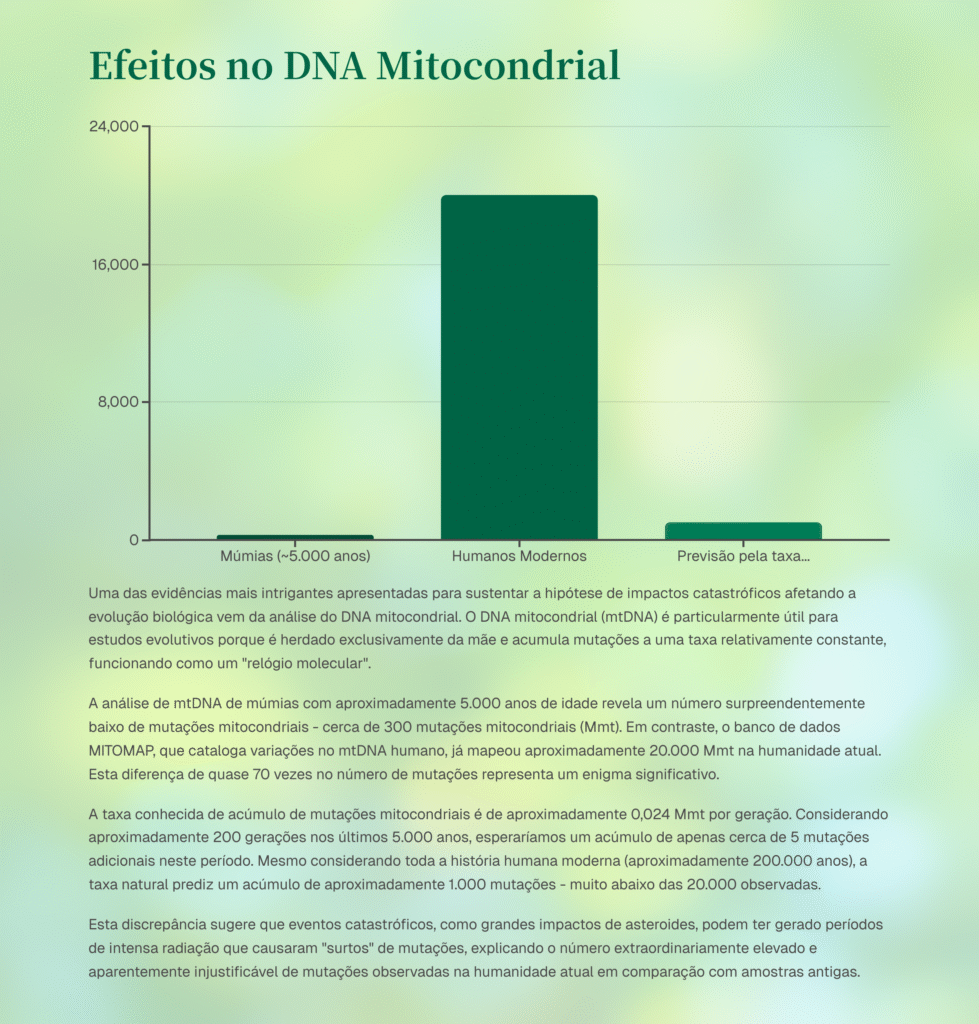
O DNA Mitocondrial: Um Caso Alarmante
O DNA mitocondrial humano (mtDNA) contém apenas 37 genes e 16.500 pares de bases em seus 33.000 nucleotídeos. Apesar de sua pequena dimensão, o banco de dados www.mitomap.org já registrou mais de 20.000 mutações deletérias nele, sendo 93,5% destas na forma de SNPs ou SNVs (mutação de um único nucleotídeo).
Este quadro revela que o mtDNA encontra-se quase totalmente estragado e degenerado. À medida que estas mutações se espalham na humanidade através das linhagens maternas, representam uma ameaça crescente à saúde populacional, podendo levar à extinção caso não sejam desenvolvidas técnicas de correção como CRISPR-Cas9.
Implicações para Cruzamentos Futuros
Presente: Alta Carga Mutacional
Atualmente, cada indivíduo carrega milhares de mutações deletérias, muitas das quais são mascaradas por heterozigose.
Futuro Próximo: Cruzamentos Perigosos
Com o acúmulo contínuo de mutações, os cruzamentos se tornarão cada vez mais arriscados, lembrando o cenário do filme “GATTACA”.
Futuro Distante: Risco de Extinção
Se a tendência continuar sem intervenção genética, a humanidade enfrentará crescente risco de extinção por colapso genético.
O Pico de Mutações: 5.000-10.000 Anos Atrás
Evidências Genômicas
Estudos em larga escala de 6.515 exomas humanos, conduzidos por Fu et al. (2013) e publicados na Nature, revelaram um achado surpreendente: aproximadamente 73% de todas as variantes codificantes de proteínas surgiram nos últimos 5.000-10.000 anos.
Esta descoberta indica uma “explosão” recente de mutações deletérias, sugerindo que eventos catastróficos no passado recente aceleraram dramaticamente as taxas mutacionais.
Convergência com Registros Históricos
Notavelmente, este período coincide com registros arqueológicos e históricos de catástrofes globais, incluindo os relatos de mais de 270 fontes antigas sobre um dilúvio global e eventos associados a rochas incandescentes.
Gerald Crabtree: Nosso Frágil Intelecto

Gerald Crabtree, professor de Stanford com índice Scopus de 121, publicou na revista Trends uma tese revolucionária intitulada “Our Fragile Intellect” (Nosso Frágil Intelecto). Nela, Crabtree demonstra que o cérebro humano está se deteriorando a passos largos.
“Entre 2 a 6.000 anos atrás, o homem era extremamente mais inteligente do que é hoje.”
Crabtree foi quem quebrou o dogma de que 1 gene produzia apenas 1 proteína em 1982, ao descobrir que fatores de transcrição podem regular a expressão gênica de forma complexa, precedendo as descobertas do splicing alternativo e controles epigenéticos.
https://pt.wikipedia.org/wiki/Our_Fragile_Intellect
Declínio Cerebral Humano
Perda de Volume Cerebral
Perda média de 10% do tamanho cerebral nos últimos 3.000 anos, documentada através de análises antropológicas comparativas.
Impacto por Mutação
Cada mutação prejudica em média 120 conexões neurais, amplificando dramaticamente o efeito de defeitos genéticos no cérebro.
Redução da Saúde Global
Estudo da Lancet documenta redução de 11% na saúde geral da população mundial entre 1990 e 2010.
A Fragilidade do DNA
Complexidade e Vulnerabilidade
O DNA humano contém 3 bilhões de nucleotídeos organizados em sequências extremamente específicas, codificando informações para produzir 5 a 6 milhões de proteínas diferentes. Esta ordenação precisa torna o DNA extraordinariamente vulnerável a desarranjos.
As forças que unem as bilhões de bases do DNA são extremamente frágeis e requerem reparo constante. Sistemas complexos de verificação, reparo e correção trabalham continuamente, mas na velhice não são suficientes para manter a qualidade de vida e saúde.
Meia-Vida Curta do DNA
Ligações Frágeis
As ligações químicas que mantêm a estrutura do DNA são relativamente fracas, tornando-o suscetível a danos por radiação, radicais livres e outros agentes.
Incompatível com Milhões de Anos
A fragilidade intrínseca do DNA torna altamente improvável sua preservação por milhões de anos, contradizendo datações geocronológicas convencionais.
Eventos Catastróficos
Momentos de alta radioatividade, como durante impactos de asteroides, geraram picos de acúmulo de mutações que reduziram ainda mais a meia-vida efetiva do DNA.
Acúmulo de Mutações no Envelhecimento Individual
O processo de envelhecimento individual demonstra claramente a entropia genética em ação. Aos 60 anos de idade, as células da pele humana acumulam aproximadamente 40.000 mutações deletérias, resultando na deformação e perda de função característica do envelhecimento.
Este fenômeno a nível individual – chamado de Entropia Genética Individual (EGI) – reflete o que ocorre em escala populacional através das gerações sucessivas, denominado Entropia Genética Populacional (EGP). Ambos os processos demonstram a tendência universal de degradação da informação genética ao longo do tempo.
Diabetes e Doenças em Expansão Exponencial
O diabetes apresentou aumento exponencial de 50% nos últimos 10 anos, com fatores causais genéticos e proteômicos atuando em conjunto com fatores ambientais e de estilo de vida. Este padrão se repete em múltiplas doenças, indicando uma tendência degenerativa global na saúde humana.
O Stress Endogâmico: Japão e Raças Puras
Homozigose Populacional
A população japonesa apresenta alta homozigose – semelhança genética entre indivíduos – devido ao histórico isolamento populacional. Esta condição gera stress endogâmico significativo.
Estudos sugerem que, sem cruzamentos com outras etnias, a população japonesa poderia estar fadada à extinção devido ao acúmulo de mutações deletérias em pool gênico empobrecido.
O Problema dos Cães de Raça
Cachorro de raça pura é sempre frágil em comparação com os vira-latas. Por quê? Porque tem muita homozigose, pobreza genética.
Para se tornar mais resistente, precisa cruzar com outras raças, aumentando a heterozigose e o vigor genético – fenômeno conhecido como “vigor híbrido”.
Melhoramento Genético: Resgatando o Passado
O melhoramento genético de plantas não cria características novas – ele resgata e recombina características que já existiam no pool gênico ancestral, mas foram perdidas através de deriva genética, seleção e mutações deletérias.
Agricultores e cientistas realizam infinitos cruzamentos para tentar recuperar um pouco das plantas ancestrais gigantes encontradas no registro fóssil. O processo envolve reunir genes derivados, não eliminados pela seleção, não mutados, evitando ao máximo os genes “gambiarrados” por adaptações degenerativas.
Plantas Gigantes do Registro Fóssil
Samambaias Gigantes
O registro fóssil apresenta samambaias com dimensões colossais, incomparáveis às espécies modernas, indicando condições ambientais e genéticas superiores.
Lepidodendron
Árvores de escama que alcançavam 30-50 metros de altura, com raízes gigantescas (stigmaria) amplamente documentadas nas camadas carboníferas.
Calamites
Cavalinhas gigantes que atingiam 30 metros de altura, contrastando drasticamente com as espécies modernas que raramente ultrapassam 1 metro.
O Contraste Entre Passado e Presente
Espécies Ancestrais Fósseis
- Plantas gigantes com sistemas radiculares extensos
- Animais de dimensões colossais (megafauna)
- Alta longevidade evidenciada em anéis de crescimento
- Menor número de patologias ósseas
- Robustez estrutural superior
Espécies Modernas Degeneradas
- Plantas anãs com sistemas radiculares reduzidos
- Animais de tamanho reduzido (exceto raras exceções)
- Longevidade drasticamente reduzida
- Alta incidência de doenças genéticas
- Fragilidade estrutural aumentada
Atmosfera Ancestral: Maior Concentração de Oxigênio
A mudança drástica no tamanho dos seres vivos indica mudança drástica de ambiente. O registro fóssil sugere que a Terra possuía atmosfera com maior concentração de oxigênio, favorecendo formas de vida gigantescas, maior longevidade e melhor controle de patógenos.
A oxigenação é amplamente citada na literatura científica como geradora de múltiplos efeitos benéficos à saúde. Técnicas como câmaras hiperbáricas, ventiladores e ozonioterapias são defendidas como ferramentas terapêuticas baseadas neste princípio.
Ozonioterapia: Evidências dos Benefícios do Oxigênio
Efeitos Antivirais
A ozonioterapia demonstrou eficácia contra diversos vírus, incluindo aplicações promissoras no tratamento de COVID-19, com mais de 3.500 artigos no PubMed.
Melhora Circulatória
Promove oxigenação tecidual, combate hipertensão e melhora a perfusão sanguínea em múltiplos sistemas orgânicos.
Ação Anti-inflamatória
Reduz processos inflamatórios sistêmicos e demonstra benefícios em condições como diabetes e doenças autoimunes.
264 Doenças Tratáveis
Desde a patente de Tesla em 1896, a técnica demonstrou aplicabilidade no tratamento de 264 condições diferentes, incluindo doenças de pele e metabólicas.
A Terra Era Mais Propícia à Vida
Evidências Convergentes
Múltiplas linhas de evidência sugerem que a Terra primitiva era significativamente mais propícia à vida:
- Atmosfera com maior concentração de oxigênio
- Menor radioatividade ambiental (ausência de Carbono-14)
- Ausência ou menor carga de patógenos virais e bacterianos
- Condições climáticas mais estáveis
- Pool gênico sem mutações deletérias acumuladas
Este cenário corresponde ao conceito criacionista de “paraíso genético”, onde todas as espécies foram criadas em estado de perfeição funcional.
Haplogrupos Mitocondriais e o Pico Mutacional
Os haplogrupos mitocondriais M e N originaram-se na África subsaariana e se espalharam pelo mundo há cerca de 60.000 anos segundo cronologia convencional. Estes haplogrupos ancestrais deram origem aos grupos europeus H, J e T.
O pico de mutações SNPs relatado por Fu et al. (2013) coincide temporalmente com a divergência destes haplogrupos europeus, implicando que por volta de 5.000-10.000 anos atrás, o número de mutações transmitidas por linhagem materna aumentou acentuadamente na Eurásia.
Este padrão sugere um evento catastrófico global que acelerou dramaticamente as taxas mutacionais, consistente com registros históricos de catástrofes planetárias.
Granitos Radioativos e Formação de SNPs
Evidências Geológicas
Granitos da crosta continental na borda das plataformas que separaram os continentes revelam níveis altos de radioatividade. Esta descoberta implica larga escala de formação de mutações na forma de SNPs e SNVs em todos os seres vivos.
A radioatividade foi particularmente prejudicial ao DNA mitocondrial germinativo descendido pelas fêmeas que viveram no momento exato das catástrofes envolvendo eventos radioativos.
Explicando as Três Evas
Este fenômeno explica porque as 3 linhagens matriarcais da humanidade (as “três Evas”), por mais que fossem contemporâneas, carregam alta diferença mutacional em seus respectivos mtDNAs.
As diferenças não refletem tempo evolutivo, mas exposição diferencial a eventos radioativos catastróficos.
Stress Endogâmico e Formação das Etnias
População Inicial Reduzida
Após evento catastrófico global, populações humanas foram drasticamente reduzidas, forçando endogamia temporária.
Gargalo Genético
Grupos isolados experimentaram forte deriva genética, fixando características específicas que definiram biotipos étnicos.
Diferenciação Rápida
Em poucas gerações, surgiram as etnias distintas: negros, caucasianos, índios mongoloides e asiáticos, cada um com características genéticas próprias.
Expansão e Diversificação
Subsequente expansão populacional e fluxo gênico geraram a diversidade atual, mas dentro dos limites estabelecidos pelos gargalos iniciais.
88 Milhões de Mutações: Implicações Temporais
O genoma humano atual contém aproximadamente 88 milhões de mutações documentadas. Com taxa média de 152 mutações deletérias por geração (25 anos), podemos calcular o tempo necessário para acumular este número:
No entanto, este cálculo assume taxas constantes. Com picos mutacionais documentados entre 5.000-10.000 anos atrás, o acúmulo foi muito mais rápido em períodos específicos, sugerindo que a pureza genética humana existiu há apenas 6.000-12.000 anos.
Cálculo do Dr. Marcos Eberlin
Metodologia Científica
O Dr. Marcos Eberlin, bioquímico brasileiro com índice Scopus 73+5, índice h de 87, e citado 34.322 vezes na literatura científica, realizou cálculo fascinante unindo as taxas mutacionais conhecidas com os picos percebidos.
Dividindo o número total de mutações identificadas no genoma humano (88 milhões) pela taxa de acúmulo por geração (considerando os picos), chegou-se à conclusão de que há apenas 6.000-12.000 anos, ou aproximadamente 10.000 anos, nós tínhamos pureza genética.
Confirmação Bíblica
Esta datação confirma o relato arqueológico de Gênesis quando fala dos ancestrais iniciais Adão e Eva, bem como genealogias estatísticas que apontam aproximadamente 6.000 anos de distância temporal dos patriarcas ancestrais da humanidade.
O MRCA: Ancestral Comum Mais Recente
Desde 2004, estudos genéticos admitem que o MRCA (Most Recent Common Ancestor – ancestral comum mais recente) de todos os humanos atuais viveu apenas alguns milhares de anos atrás, não milhões de anos como pregava a teoria evolucionista tradicional.
“O MRCA de todos os humanos atuais viveu apenas alguns milhares de anos atrás.” – Rohde, Olson & Chang, Nature (2004)
Esta descoberta revolucionária, publicada na prestigiosa revista Nature, alinha-se perfeitamente com as cronologias bíblicas e desafia fundamentalmente as narrativas evolucionistas de milhões de anos de história humana.
Tecidos Moles em Fósseis: Refutando Milhões de Anos
Descoberta Revolucionária
Mary Schweitzer descobriu tecidos ainda orgânicos, incluindo vasos sanguíneos flexíveis e células, em ossos de Tiranossauro Rex supostamente datados em 68 milhões de anos.
Esta descoberta gerou imenso embaraço para a comunidade científica evolucionista, pois tecidos orgânicos não podem resistir à ação de bactérias e fungos por mais de alguns milhares de anos, no máximo.
Mais de 1.200 Casos
Atualmente, mais de 1.200 fósseis foram documentados contendo tecidos orgânicos originais, incluindo:
- DNA preservado
- 17 tipos de biomoléculas originais
- Colágeno com elasticidade
- Vasos sanguíneos flexíveis
- Células com núcleos visíveis
Reações de Fenton: A “Porcaria” Científica
Para tentar explicar a preservação impossível de tecidos orgânicos por milhões de anos, Mary Schweitzer e outros publicaram artigos defendendo que “reações de Fenton” com óxido de ferro teriam preservado milagrosamente os tecidos por 68 milhões de anos.
O Dr. Marcos Eberlin, que possui duas publicações analisando reações de Fenton, classificou estas publicações – incluindo da prestigiosa revista Science – como “porcaria”. Eberlin argumenta que experimentos de apenas 2 anos não podem justificar preservação de 68 milhões de anos, e que as reações propostas não têm capacidade de preservar tecidos complexos por períodos tão extensos.
Muitos cientistas preferem questionar o “dogma” de que proteínas e DNA não se preservam por muito tempo, do que questionar as datações “inerrantes” e “absolutas”, porque isso representa comprar imensa briga com a patrulha darwinista.
Carbono-14 em Diamantes: Destruindo Bilhões de Anos
Descoberta Devastadora
O geofísico John Baumgardner e colaboradores testaram diamantes de múltiplas fontes, incluindo do manto terrestre, supostamente com bilhões de anos de idade. Surpreendentemente, encontraram Carbono-14 datável em quantidade significativa.
O problema: Carbono-14 tem meia-vida de apenas 5.730 anos, e não deveria estar presente após 100.000-250.000 anos no máximo. Sua presença em diamantes “antigos” é impossível dentro da cronologia evolucionista.
Diamantes Praticamente Incontamináveis
Diamantes são estruturas cristalinas extremamente resistentes à contaminação externa. O Carbono-14 encontrado é original, não contaminação posterior.
Esta descoberta “rasga” literalmente os bilhões de anos da geocronologia convencional.
Datações Radiométricas: O Grande Engano
Pressuposto Fatal: Constância de Decaimento
Toda a geocronologia se baseia no pressuposto de que as taxas de decaimento radioativo permaneceram constantes ao longo de bilhões de anos.
Evidências de Aceleração
Múltiplos estudos demonstram que o decaimento nuclear pode ser acelerado por plasma, efeito piezoelétrico, temperatura, pressão e outros fatores.
Impactos de Asteroides
Eventos catastróficos como impactos de asteroides geram condições extremas que aceleram dramaticamente o decaimento radioativo.
Aparência de Idade
Rochas podem adquirir “aparência de idade” artificialmente alta quando submetidas a eventos que aceleram o decaimento durante períodos curtos.
Colapso da Geocronologia
A descoberta de que o decaimento não foi constante destrói completamente a confiabilidade das datações radiométricas de longa escala temporal.
Tokamaks e Fusão Nuclear: Lições para Geologia
Máquinas Tokamak, desenvolvidas para fusão nuclear controlada, revelaram que acelerando suficientemente os elétrons através de plasma em alta temperatura, é possível atingir o núcleo atômico e alterar a composição nuclear.
Impactos de asteroides e eventos tectônicos violentos geram condições similares: plasma, efeito piezoelétrico (pressão extrema em cristais), temperaturas altíssimas e ondas sonoras intensas. Estas condições podem arrancar nêutrons e prótons dos núcleos atômicos, acelerando o decaimento.
Isto implica que durante catástrofes geológicas, rochas poderiam “envelhecer” artificialmente em períodos muito curtos, adquirindo aparência de milhões ou bilhões de anos quando na verdade têm apenas milhares.
Uniformitarismo de Lyell: O Erro de Darwin
A Aposta Errada
Charles Darwin baseou sua teoria no uniformitarismo de Charles Lyell, que propunha que processos geológicos sempre ocorreram nas mesmas taxas que observamos hoje – “o presente é a chave do passado”.
Darwin desprezou os geólogos fundadores da disciplina e adotou as ideias de Lyell, um advogado sem formação geológica, justamente porque o uniformitarismo fornecia os milhões de anos necessários para sua teoria evolutiva.
Geologia Moderna Rejeita Uniformitarismo
A geologia moderna abandonou o uniformitarismo de Lyell. As evidências de catástrofes globais são esmagadoras:
- Camadas sedimentares globais extensas
- Quatrilhões de pedras despedaçadas misturadas ao barro
- Bolders gigantes arrastados
- Fósseis completos sepultados rapidamente
Catastrofismo: A Realidade Geológica
Camadas Globais
Camadas sedimentares grossas, largas e extensas que cobrem continentes inteiros só podem ser formadas por catástrofes de escala global, não por processos lentos e graduais.
Sepultamento Rápido
Para fossilizar um animal continental gigante completo, é necessária maré altíssima de água e lama varrendo o continente rapidamente.
Ausência de Fossilização Moderna
Não vemos elefantes, girafas ou rinocerontes sendo fossilizados hoje porque os eventos catastróficos que formaram as camadas geológicas não ocorrem atualmente.
LPI e Separação Continental
A fenda meso-oceânica é uma gigantesca LPI (Large Igneous Province), evidenciando separação continental violenta e catastrófica, não gradual.
Estratificação Espontânea
Experimentos laboratoriais demonstram que misturas de partículas de diferentes tamanhos e densidades se segregam espontaneamente quando submetidas a fluxos turbulentos de água, formando camadas distintas sem necessidade de milhões de anos.
Este fenômeno, chamado “estratificação espontânea” ou “segregação automática”, explica perfeitamente a formação das camadas sedimentares do registro geológico através de eventos catastróficos envolvendo grandes volumes de água em movimento.
As camadas não representam períodos geológicos sucessivos, mas fases de deposição durante um único evento catastrófico global, organizadas por propriedades físico-químicas do material erodido.
O Paradoxo da Estase Morfológica
O Problema para o Evolucionismo
Stephen Jay Gould e Niles Eldredge documentaram o “paradoxo da estase morfológica”: formas fósseis aparecem subitamente no registro, permanecem essencialmente inalteradas por supostos milhões de anos, e depois desaparecem ou persistem até hoje como “fósseis vivos”.
Este padrão contradiz completamente a expectativa evolutiva de mudança gradual e constante. Como Ernst Mayr admitiu: “Esta constância de formas de vida em face de genomas tremendamente dinâmicos é um dos maiores problemas da biologia evolutiva contemporânea.”
Especiação Rápida vs. Estase Fóssil
Observamos especiação acontecendo em tempo real e histórico-arqueológico (centenas de anos), mas no registro fóssil vemos repetição de mesmas formas por “milhões de anos”.
A explicação mais parcimoniosa: os fósseis não representam milhões de anos, mas sim populações sepultadas rapidamente durante catástrofe global.
Fósseis Vivos: 4.229 Gêneros Inalterados
Nautilus
Molusco cefalópode com concha espiralada, praticamente idêntico aos fósseis de “centenas de milhões de anos”.
Límulus (Caranguejo-Ferradura)
Considerado inalterado por “450 milhões de anos”, desafia completamente a teoria da evolução.
Ginkgo Biloba
Árvore com folhas características idênticas aos fósseis “jurássicos”, sem mudança morfológica detectável.
Celacanto
Peixe “extinto há 65 milhões de anos” redescoberto vivo em 1938, praticamente idêntico aos fósseis.
Atualmente contamos com 4.229 gêneros de “fósseis vivos” – organismos morfologicamente idênticos aos seus supostos ancestrais de milhões de anos atrás. Esta estase morfológica massiva é inexplicável pela teoria evolucionista, mas perfeitamente consistente com criação recente seguida de degradação.
Templates Fósseis vs. Templates Biológicos
Apesar da enorme diferença no número total de espécies (250.000-300.000 fósseis vs. 2-10 milhões de espécies vivas), o número de arquétipos ou “templates” biológicos básicos permanece praticamente igual.
Esta observação crucial revela que a biomodificação evolutiva está restrita a variações dentro de grupos estabelecidos (tipos básicos ancestrais), não havendo surgimento de novos arquétipos fundamentais. A diversidade ocorre dentro de limites, exatamente como previsto pelo modelo criacionista.
Surgimento Súbito de Arquétipos no Registro Fóssil
Um dos fatos mais embaraçosos para a teoria da evolução é o surgimento “pronto” e súbito de formas de vida complexas no registro fóssil, fenômeno mais dramático na “Explosão Cambriana”.
Milhares de paleontólogos, liderados por Gould e Eldredge, documentaram que os principais arquétipos biológicos aparecem abruptamente, completamente formados, sem ancestrais transicionais graduais.
“A explosão cambriana foi um evento evolutivo de grande magnitude intimamente ligado à origem dos animais” – admissão de que o padrão fóssil contradiz gradualismo darwiniano. – Antcliffe et al. (2014)
Baraminologia: Limites da Ancestralidade Comum
Descontinuidade Sistemática
A baraminologia é o estudo dos limites naturais entre grupos de organismos. Pesquisadores como Kurt Patrick Wise (PhD em paleontologia por Harvard, orientado por Stephen Jay Gould) documentam que existem descontinuidades claras entre grupos.
Plantas cruzam entre espécies, gêneros, até o nível de família em enxertos. Passando este limite, não há cruzamentos viáveis, demonstrando que o nível “família” aproximadamente define os limites da ancestralidade comum.
Tipos Básicos Criados
A evidência sugere que existiram tipos básicos originais (baramins), criados prontos com grande diversidade potencial em seus genomas.
A diversificação subsequente ocorreu dentro destes limites estabelecidos, gerando as milhões de espécies atuais através de variação, não através de transformação ilimitada de um tipo em outro completamente diferente.
Complexidade Irredutível: Michael Behe
Michael Behe, bioquímico da Universidade Lehigh, introduziu o conceito de “complexidade irredutível” em seu livro “A Caixa Preta de Darwin”. Sistemas irredutivelmente complexos são aqueles que requerem múltiplos componentes funcionando em conjunto para ter qualquer função.
O exemplo clássico é o flagelo bacteriano – um “motor” molecular rotativo com múltiplas peças precisamente coordenadas. Remova qualquer componente e o sistema perde completamente sua função. Como tal sistema poderia evoluir gradualmente se estágios intermediários são não-funcionais e, portanto, não selecionáveis?
Behe argumenta que sistemas irredutivelmente complexos requerem design inteligente, não podendo surgir por processos darwinianos graduais.
James Tour: “Aminoácidos Não Se Ligam Sozinhos”
Autoridade em Química Sintética
James Tour é considerado um dos maiores químicos sintéticos vivos, com centenas de publicações e múltiplas patentes. Sua expertise em síntese molecular lhe dá autoridade única para avaliar alegações sobre origem da vida.
Tour afirma categoricamente que aminoácidos não se ligam espontaneamente para formar proteínas funcionais nas condições propostas pela hipótese da “sopa primordial”. A química simplesmente não funciona da maneira que os cenários evolutivos requerem.
Suas críticas devastadoras às hipóteses de origem da vida expõem a enorme lacuna entre demonstrações químicas reais e especulações teóricas.
O Fracasso da Hipótese da Sopa Primordial
Problema 1: Formação de Aminoácidos
Experimentos como Miller-Urey produzem apenas misturas racêmicas de aminoácidos simples, não as formas quirais específicas necessárias para a vida.
Problema 2: Polimerização
Aminoácidos não se ligam espontaneamente em água para formar proteínas – a reação é termodinamicamente desfavorável.
Problema 3: Sequência Específica
Mesmo que proteínas pudessem formar, proteínas funcionais requerem sequências específicas extremamente improváveis de surgir ao acaso.
Problema 4: Informação
DNA/RNA codifica informação especificada – um conceito completamente ausente em processos químicos não-dirigidos.
Problema 5: Sistema Integrado
A vida requer DNA, RNA, proteínas e membranas funcionando em sistema integrado. Nenhum componente isolado tem função.
Movimento do Design Inteligente
Mais de 10% dos Cientistas
Pesquisas recentes indicam que mais de 10% dos cientistas, muitos deles de áreas relacionadas à biologia, geologia e paleontologia, se posicionaram publicamente em favor do Design Inteligente.
Uma lista de protesto com milhares de cientistas com PhD, incluindo a “Dissent from Darwin” com mais de 1.000 assinaturas, clama por reforma na ciência e questiona dogmas darwinianos.
Perseguição Acadêmica
O documentário “Expelled: No Intelligence Allowed” (2008), que arrecadou mais de 7,7 milhões de dólares, documenta a intensa perseguição que cientistas sofrem quando questionam o darwinismo ou sugerem design.
Casos incluem:
- David Coppedge, demitido da NASA após 14 anos
- Guillermo Gonzalez, negado tenure apesar de qualificações
- Richard Sternberg, assediado no Smithsonian
Coreia do Sul: Capital Mundial do Criacionismo
A Coreia do Sul, líder mundial em educação e tecnologia, é frequentemente chamada de “capital do criacionismo”. Considerando sua população, é o país campeão isolado em patentes no mundo.
Esta realidade desafia completamente a narrativa de que aceitar criacionismo gera “atraso científico”. Pelo contrário, a Coreia do Sul demonstra que excelência científica e tecnológica é perfeitamente compatível com rejeição do darwinismo.
O país possui educação avançada, indústria tecnológica pujante e produção científica robusta, simultaneamente mantendo forte tradição criacionista. Este fato empírico refuta o argumento de que ensinar criacionismo prejudicaria a educação.
CRISPR-Cas9: Corrigindo a Degeneração
Revolução na Edição Genética
A tecnologia CRISPR-Cas9, desenvolvida por Jennifer Doudna e Emmanuelle Charpentier (Prêmio Nobel 2020), fornece método rápido, barato e extremamente preciso de alterar sequências de DNA.
Esta tecnologia oferece esperança de corrigir mutações deletérias acumuladas ao longo de gerações. Pesquisadores já usaram CRISPR em modelos pré-clínicos para corrigir mutações que causam:
- Distrofia muscular de Duchenne
- Fibrose cística
- Anemia falciforme
- Diversas outras doenças genéticas
Ironicamente, a necessidade de edição genética valida a teoria da degeneração – não estaríamos corrigindo mutações se elas representassem “evolução” benéfica.
O Futuro: Regeneração ou Extinção?
Cenário 1: Extinção por Colapso Genético
Se as taxas atuais de acúmulo de mutações deletérias continuarem sem intervenção, a humanidade enfrentará crescente carga de doenças genéticas até eventual extinção.
Cenário 2: Correção Tecnológica
Tecnologias como CRISPR podem corrigir sistematicamente mutações deletérias, revertendo parcialmente a degeneração genética e estendendo a viabilidade populacional.
Cenário 3: Regeneração Divina
Perspectiva bíblica aponta para futura regeneração sobrenatural de todas as coisas, restaurando a criação à sua condição original perfeita.
Conclusões: Paradigma da Degeneração
Evidências Convergentes
Múltiplas linhas de evidência científica convergem para o mesmo paradigma fundamental: as espécies não estão evoluindo para maior complexidade, mas degenerando a partir de genomas originalmente perfeitos:
- Proporção Mutacional: 1 milhão de mutações deletérias para cada benéfica (E. coli)
- Acúmulo Documentado: 88 milhões de variantes no genoma humano, 73% surgidas recentemente
- Pico Mutacional: Explosão de mutações entre 5.000-10.000 anos atrás
- Motores Degenerativos: Seleção natural, deriva e mutações empobrecem o pool gênico
- Declínio Cognitivo: Perda de 10% do volume cerebral em 3.000 anos
- Doenças Crescentes: Aumento exponencial de doenças genéticas globalmente
- Estase Fóssil: Ausência de evolução morfológica no registro geológico
- Tecidos Preservados: 1.200+ fósseis com tecidos orgânicos refutam milhões de anos
- C14 em Diamantes: Datações radiométricas demonstradas falsas
- Pureza Recente: Cálculos indicam genoma perfeito há 6.000-12.000 anos
Implicações Finais
Revolução Científica Necessária
As evidências apresentadas exigem mudança fundamental de paradigma. A Teoria da Degeneração das Espécies (TDE) oferece estrutura mais coerente e parcimoniosa para compreender:
- O melhoramento genético como recuperação, não criação
- A necessidade de cruzamentos para vigor híbrido
- O contraste entre gigantismo fóssil e miniaturização moderna
- A urgência de técnicas como CRISPR para correção genética
- A realidade de criação recente seguida de degeneração
Chamado à Ação A comunidade científica deve:
-
Abandonar dogmatismo evolucionista
-
Reavaliar cronologias baseadas em datações falhas
-
Reconhecer evidências de design inteligente
-
Investir em correção genética urgente
-
Restaurar diálogo honesto sobre origens
“A verdade científica não é determinada por consenso, mas por correspondência com a realidade observável.”
Referências
-
↑ Andersson, Anna-Maria; Jensen, Tina K.; Juul, Anders; Petersen, Jørgen H.; Jørgensen, Torben; Skakkebæk, Niels E. (1 de dezembro de 2007). «Secular Decline in Male Testosterone and Sex Hormone Binding Globulin Serum Levels in Danish Population Surveys». The Journal of Clinical Endocrinology & Metabolism (12): 4696–4705. ISSN 0021-972X. doi:10.1210/jc.2006-2633. Consultado em 11 de outubro de 2025
-
↑ Chodick, Gabriel; Epstein, Shdema; Shalev, Varda (9 de março de 2020). «Secular trends in testosterone- findings from a large state-mandate care provider». Reproductive Biology and Endocrinology (1). ISSN 1477-7827. doi:10.1186/s12958-020-00575-2. Consultado em 11 de outubro de 2025
-
↑ Santi, Daniele; Spaggiari, Giorgia; Furini, Chiara; Griseta, Valentina; Zizzi, Eric A.; Granata, Antonio R.M.; Simoni, Manuela (2024). «Secular Trends in Serum Testosterone and Luteinizing Hormone Levels Indicate an Ongoing Resetting of Hypothalamic-Pituitary-Gonadal Function in Healthy Men». doi.org. Consultado em 11 de outubro de 2025
-
↑ Travison, Thomas G.; Araujo, Andre B.; O’Donnell, Amy B.; Kupelian, Varant; McKinlay, John B. (1 de janeiro de 2007). «A Population-Level Decline in Serum Testosterone Levels in American Men». The Journal of Clinical Endocrinology & Metabolism (1): 196–202. ISSN 0021-972X. doi:10.1210/jc.2006-1375. Consultado em 11 de outubro de 2025
-
↑ Levine, Hagai; Jørgensen, Niels; Martino-Andrade, Anderson; Mendiola, Jaime; Weksler-Derri, Dan; Mindlis, Irina; Pinotti, Rachel; Swan, Shanna H. (1 de novembro de 2017). «Temporal trends in sperm count: a systematic review and meta-regression analysis». Human Reproduction Update (6): 646–659. ISSN 1460-2369. PMC 6455044
 . PMID 28981654. doi:10.1093/humupd/dmx022. Consultado em 11 de outubro de 2025
. PMID 28981654. doi:10.1093/humupd/dmx022. Consultado em 11 de outubro de 2025 -
↑ Levine, Hagai; Jørgensen, Niels; Martino-Andrade, Anderson; Mendiola, Jaime; Weksler-Derri, Dan; Jolles, Maya; Pinotti, Rachel; Swan, Shanna H (15 de novembro de 2022). «Temporal trends in sperm count: a systematic review and meta-regression analysis of samples collected globally in the 20th and 21st centuries». Human Reproduction Update (2): 157–176. ISSN 1355-4786. doi:10.1093/humupd/dmac035. Consultado em 11 de outubro de 2025
-
1 2 3 Sanford, John; Brewer, Wesley; Smith, Franzine; Baumgardner, John (17 de setembro de 2015). «The waiting time problem in a model hominin population». Theoretical Biology and Medical Modelling (em inglês) (1). 18 páginas. ISSN 1742-4682. PMC 4573302
 . PMID 26376851. doi:10.1186/s12976-015-0016-z. Consultado em 24 de outubro de 2020
. PMID 26376851. doi:10.1186/s12976-015-0016-z. Consultado em 24 de outubro de 2020 -
1 2 Sanford, John C.; Baumgardner, John R.; Brewer, Wesley H. (10 de abril de 2013). «Selection Threshold Severely Constrains Capture of Beneficial Mutations». WORLD SCIENTIFIC: 264–297. ISBN 9789814508711. doi:10.1142/9789814508728_0011
-
1 2 Basener, William F.; Sanford, John C. (junho de 2018). «The fundamental theorem of natural selection with mutations». Journal of Mathematical Biology (7): 1589–1622. ISSN 1432-1416. PMC 5906570
 . PMID 29116373. doi:10.1007/s00285-017-1190-x. Consultado em 5 de abril de 2021
. PMID 29116373. doi:10.1007/s00285-017-1190-x. Consultado em 5 de abril de 2021 -
1 2 Basener, William F. (10 de abril de 2013). «Limits of Chaos and Progress in Evolutionary Dynamics». WORLD SCIENTIFIC: 87–104. ISBN 978-981-4508-71-1. doi:10.1142/9789814508728_0004. Consultado em 17 de dezembro de 2022
-
↑ Ninio, Jacques (7 de maio de 1984). «The Neutral Theory of Molecular Evolution». FEBS Letters (1): 210–211. ISSN 0014-5793. doi:10.1016/0014-5793(84)81411-8. Consultado em 27 de julho de 2023
-
↑ Novick, Rose (16 de fevereiro de 2023). «The neutral theory of conceptual complexity». Philosophy of Science (em inglês): 1–10. ISSN 0031-8248. doi:10.1017/psa.2023.25. Consultado em 27 de julho de 2023
-
1 2 3 The 1000 Genomes Project Consortium; Corresponding authors; Auton, Adam; Abecasis, Gonçalo R.; Steering committee; Altshuler, David M.; Durbin, Richard M.; Abecasis, Gonçalo R.; Bentley, David R. (1 de outubro de 2015). «A global reference for human genetic variation». Nature (em inglês) (7571): 68–74. ISSN 0028-0836. PMC 4750478
 . PMID 26432245. doi:10.1038/nature15393. Consultado em 30 de junho de 2023
. PMID 26432245. doi:10.1038/nature15393. Consultado em 30 de junho de 2023 -
1 2 3 Conrad, Donald F; Keebler, Jonathan E M; DePristo, Mark A; Lindsay, Sarah J; Zhang, Yujun; Casals, Ferran; Idaghdour, Youssef; Hartl, Chris L; Torroja, Carlos (julho de 2011). «Variation in genome-wide mutation rates within and between human families». Nature Genetics (em inglês) (7): 712–714. ISSN 1546-1718. doi:10.1038/ng.862. Consultado em 1 de julho de 2023
-
1 2 Berthault, G. (1 de setembro de 2002). «Analysis of Main Principles of Stratigraphy on the Basis of Experimental Data». Lithology and Mineral Resources (em inglês) (5): 442–446. ISSN 1608-3229. doi:10.1023/A:1020220232661. Consultado em 12 de abril de 2022
-
1 2 Berthault, G. (1 de setembro de 2002). «Analysis of Main Principles of Stratigraphy on the Basis of Experimental Data». Lithology and Mineral Resources (em inglês) (5): 442–446. ISSN 1608-3229. doi:10.1023/A:1020220232661. Consultado em 12 de abril de 2022
-
1 2 Shormann, David (7 de outubro de 2020). «The Giant 1912 Eruption of Novarupta-Katmai: Laboratory Illustrating Earth's Catastrophic Past». Proceedings of the International Conference on Creationism (1). ISSN 2639-4006. Consultado em 12 de abril de 2022
-
↑ Senter, Philip J. (1 de agosto de 2022). «Radiocarbon in Dinosaur Bones Revisited». The American Biology Teacher (em inglês) (6): 336–341. ISSN 0002-7685. doi:10.1525/abt.2022.84.6.336. Consultado em 12 de maio de 2024
-
↑ Senter, Philip J. (1 de maio de 2021). «Preservation of Soft Tissues in Dinosaur Fossils». The American Biology Teacher (em inglês) (5): 298–302. ISSN 0002-7685. doi:10.1525/abt.2021.83.5.298. Consultado em 12 de maio de 2024
-
↑ Gobbo, Silvia Regina; Bertini, Reinaldo J. (2014). «Tecidos moles (não resistentes): como se fossilizam?». Terrae Didatica (1): 2–13. ISSN 1980-4407. doi:10.20396/td.v10i1.8637374. Consultado em 12 de maio de 2024
-
1 2 Senter, Philip J. (11 de dezembro de 2022). «Cells and soft tissues in fossil bone: A review of preservation mechanisms, with corrections of misconceptions». Palaeontologia Electronica (em English) (3): 1–52. ISSN 1094-8074. doi:10.26879/1248. Consultado em 1 de agosto de 2023 !CS1 manut: Língua não reconhecida (link)
-
1 2 Senter, Philip J. (1 de agosto de 2022). «Radiocarbon in Dinosaur Bones Revisited». The American Biology Teacher (6): 336–341. ISSN 0002-7685. doi:10.1525/abt.2022.84.6.336. Consultado em 1 de agosto de 2023
-
↑ Gallucci, John E.; Woolslayer, Grace; Barker, Kelsey; Kibelstis, Brian; Tumarkin-Deratzian, Allison R.; Ullmann, Paul V.; Grandstaff, David E.; Terry, Dennis O. (maio de 2024). «Controls on Soft Tissue and Cellular Preservation in Late Eocene and Oligocene Vertebrate Fossils from the White River and Arikaree Groups of Nebraska, South Dakota, and Wyoming». Minerals (em inglês) (5). 497 páginas. ISSN 2075-163X. doi:10.3390/min14050497. Consultado em 12 de maio de 2024
-
1 2 Fu, Wenqing; O’Connor, Timothy D.; Jun, Goo; Kang, Hyun Min; Abecasis, Goncalo; Leal, Suzanne M.; Gabriel, Stacey; Rieder, Mark J.; Altshuler, David (janeiro de 2013). «Analysis of 6,515 exomes reveals the recent origin of most human protein-coding variants». Nature (em inglês) (7431): 216–220. ISSN 1476-4687. doi:10.1038/nature11690. Consultado em 11 de abril de 2022
-
1 2 3 4 Crabtree, Gerald R. (1 de janeiro de 2013). «Our fragile intellect. Part I». Trends in Genetics. 29 (1): 1–3. ISSN 0168-9525. doi:10.1016/j.tig.2012.10.002
-
↑ DeSilva, Jeremy M.; Traniello, James F. A.; Claxton, Alexander G.; Fannin, Luke D. (2021). «When and Why Did Human Brains Decrease in Size? A New Change-Point Analysis and Insights From Brain Evolution in Ants». Frontiers in Ecology and Evolution. ISSN 2296-701X. doi:10.3389/fevo.2021.742639/full?utm_source=yxnews&utm_medium=mobile. Consultado em 29 de julho de 2023
-
1 2 Campbell, Catarina D.; Chong, Jessica X.; Malig, Maika; Ko, Arthur; Dumont, Beth L.; Han, Lide; Vives, Laura; O'Roak, Brian J.; Sudmant, Peter H. (novembro de 2012). «Estimating the human mutation rate using autozygosity in a founder population». Nature Genetics (em inglês) (11): 1277–1281. ISSN 1546-1718. doi:10.1038/ng.2418. Consultado em 24 de julho de 2023
-
↑ DeSilva, Jeremy M.; Traniello, James F. A.; Claxton, Alexander G.; Fannin, Luke D. (2021). «When and Why Did Human Brains Decrease in Size? A New Change-Point Analysis and Insights From Brain Evolution in Ants». Frontiers in Ecology and Evolution. ISSN 2296-701X. doi:10.3389/fevo.2021.742639/full?utm_source=yxnews&utm_medium=mobile. Consultado em 29 de julho de 2023
-
↑ Basener, William F.; Sanford, John C. (2018). «The fundamental theorem of natural selection with mutations». Journal of Mathematical Biology. 76 (7): 1589–1622. ISSN 1432-1416. PMC 5906570
 . PMID 29116373. doi:10.1007/s00285-017-1190-x
. PMID 29116373. doi:10.1007/s00285-017-1190-x -
↑ Arslan, Ruben C.; Willführ, Kai P.; Frans, Emma M.; Verweij, Karin J. H.; Bürkner, Paul-Christian; Myrskylä, Mikko; Voland, Eckart; Almqvist, Catarina; Zietsch, Brendan P. (13 de setembro de 2017). «Older fathers' children have lower evolutionary fitness across four centuries and in four populations». Proceedings of the Royal Society B: Biological Sciences (1862). 20171562 páginas. PMC 5597845
 . PMID 28904145. doi:10.1098/rspb.2017.1562. Consultado em 17 de dezembro de 2022
. PMID 28904145. doi:10.1098/rspb.2017.1562. Consultado em 17 de dezembro de 2022 -
↑ Borowska, Alicja; Szwaczkowski, Tomasz; Kamiński, Stanisław; Hering, Dorota M.; Kordan, Władysław; Lecewicz, Marek (1 de maio de 2018). «Identification of genome regions determining semen quality in Holstein-Friesian bulls using information theory». Animal Reproduction Science (em inglês). 192: 206–215. ISSN 0378-4320. doi:10.1016/j.anireprosci.2018.03.012
-
↑ Arslan, Ruben C.; Willführ, Kai P.; Frans, Emma M.; Verweij, Karin J. H.; Bürkner, Paul-Christian; Myrskylä, Mikko; Voland, Eckart; Almqvist, Catarina; Zietsch, Brendan P. (13 de setembro de 2017). «Older fathers' children have lower evolutionary fitness across four centuries and in four populations». Proceedings of the Royal Society B: Biological Sciences. 284 (1862). 20171562 páginas. PMC 5597845
 . PMID 28904145. doi:10.1098/rspb.2017.1562
. PMID 28904145. doi:10.1098/rspb.2017.1562 -
↑ Casellas, J.; Varona, L. (1 de agosto de 2011). «Short communication: Effect of mutation age on genomic predictions». Journal of Dairy Science (em inglês). 94 (8): 4224–4229. ISSN 0022-0302. doi:10.3168/jds.2011-4186
-
↑ Shmookler Reis, Robert J.; Ebert, Robert H. (1 de janeiro de 1996). «Genetics of aging: Current animal models». Experimental Gerontology. In Memory of Samuel Goldstein 1938-1994 (em inglês). 31 (1): 69–81. ISSN 0531-5565. doi:10.1016/0531-5565(95)00019-4
-
1 2 Crevecoeur, Guibert U. (1 de dezembro de 2019). «Entropy growth and information gain in operating organized systems». AIP Advances. 9 (12). 125041 páginas. doi:10.1063/1.5128315
-
↑ Crevecoeur, Guibert U. (1 de dezembro de 2019). «Entropy growth and information gain in operating organized systems». AIP Advances. 9 (12). 125041 páginas. ISSN 2158-3226. doi:10.1063/1.5128315
-
↑ Brewer, Wesley H.; Baumgardner, John R.; Sanford, John C. (10 de abril de 2013). «Using Numerical Simulation to Test the ?Mutation-Count? Hypothesis». WORLD SCIENTIFIC: 298–311. ISBN 978-981-4508-71-1. doi:10.1142/9789814508728_0012
-
↑ You, Wenpeng; Henneberg, Maciej (2018). «Cancer incidence increasing globally: The role of relaxed natural selection». Evolutionary Applications (em inglês). 11 (2): 140–152. ISSN 1752-4571. PMC 5775494
 . PMID 29387151. doi:10.1111/eva.12523
. PMID 29387151. doi:10.1111/eva.12523 -
↑ Carter, Robert W; Sanford, John C (12 de outubro de 2012). «A new look at an old virus: patterns of mutation accumulation in the human H1N1 influenza virus since 1918». Theoretical Biology & Medical Modelling. 9. ISSN 1742-4682. PMC 3507676
 . PMID 23062055. doi:10.1186/1742-4682-9-42
. PMID 23062055. doi:10.1186/1742-4682-9-42 -
↑ Lu, Hongfang; Yang, Ying; Allister, Emma M.; Wijesekara, Nadeeja; Wheeler, Michael B. (1 de agosto de 2008). «The Identification of Potential Factors Associated with the Development of Type 2 Diabetes: A Quantitative Proteomics Approach». Molecular & Cellular Proteomics (em inglês) (8): 1434–1451. ISSN 1535-9476. PMID 18448419. doi:10.1074/mcp.M700478-MCP200. Consultado em 26 de setembro de 2020
-
1 2 Murray, Christopher JL; Lopez, Alan D.; Zonies, David; Zheng, Zhi-Jie; Zaidi, Anita KM; Yeh, Pon-Hsiu; Wulf, Sarah; Woolf, Anthony D.; Wolfe, Frederick (15 de dezembro de 2012). «Years lived with disability (YLDs) for 1160 sequelae of 289 diseases and injuries 1990–2010: a systematic analysis for the Global Burden of Disease Study 2010». The Lancet (em English). 380 (9859): 2163–2196. ISSN 0140-6736. PMID 23245607. doi:10.1016/S0140-6736(12)61729-2 !CS1 manut: Língua não reconhecida (link)
-
↑ Roizman, Bernard (1996). Baron, Samuel, ed. «Multiplication». Galveston (TX): University of Texas Medical Branch at Galveston. ISBN 978-0-9631172-1-2. PMID 21413311. Consultado em 20 de março de 2021
-
↑ Bergman, Jerry (1999). «Did God make pathogenic viruses? - creation.com». creation.com (em inglês). Journal of Creation 13(1):115–125, April 1999. Consultado em 20 de março de 2021
-
1 2 Dos Santos, Laurita; Rybarczyk, J.L.; Gerhardt, G.J.L. (8 de janeiro de 2011). «Triplet Entropy in H1N1 Virus». Trends in Computational and Applied Mathematics (3): 253–261. ISSN 2676-0029. doi:10.5540/tema.2011.012.03.0253. Consultado em 12 de maio de 2024
-
1 2 Pan, Keyao; Deem, Michael W. (7 de novembro de 2011). «Quantifying selection and diversity in viruses by entropy methods, with application to the haemagglutinin of H3N2 influenza». Journal of The Royal Society Interface (em inglês) (64): 1644–1653. ISSN 1742-5689. PMC 3177615
 . PMID 21543352. doi:10.1098/rsif.2011.0105. Consultado em 12 de maio de 2024
. PMID 21543352. doi:10.1098/rsif.2011.0105. Consultado em 12 de maio de 2024 -
↑ Basener, William F.; Sanford, John C. (1 de junho de 2018). «The fundamental theorem of natural selection with mutations». Journal of Mathematical Biology (em inglês) (7): 1589–1622. ISSN 1432-1416. PMC 5906570
 . PMID 29116373. doi:10.1007/s00285-017-1190-x. Consultado em 20 de março de 2021
. PMID 29116373. doi:10.1007/s00285-017-1190-x. Consultado em 20 de março de 2021 -
↑ H., Gillespie, J. Substitution Processes in Molecular Evolution. III. Deleterious Alleles. [S.l.: s.n.] OCLC 678648561
-
↑ Kondepudi, Dilip K; De Bari, Benjamin; Dixon, James A. (16 de novembro de 2020). «Dissipative Structures, Organisms and Evolution». Entropy (11). 1305 páginas. ISSN 1099-4300. doi:10.3390/e22111305. Consultado em 1 de julho de 2023
-
↑ Collier, John (1 de março de 1986). «Entropy in evolution». Biology and Philosophy (em inglês) (1): 5–24. ISSN 1572-8404. doi:10.1007/BF00127087. Consultado em 1 de julho de 2023
-
↑ Brooks, Daniel R.; Leblond, Paul H.; Cumming, D. David (julho de 1984). «Information and entropy in a simple evolution model». Journal of Theoretical Biology (1): 77–93. ISSN 0022-5193. doi:10.1016/s0022-5193(84)80112-5. Consultado em 1 de julho de 2023
-
↑ Ayala, Francisco J. (julho de 1985). «Neo-darwinism: An uneven assessment. Evolutionary theory: Paths into the future. Edited by J. W. P<scp>OLLARD</scp>. John Wiley and Sons, 1984, Pp. 271. £21.50». BioEssays (1): 44–45. ISSN 0265-9247. doi:10.1002/bies.950030113. Consultado em 1 de julho de 2023
-
↑ Costa-Cabanas, Diogo; Chalub, Fabio A. C. C.; Souza, Max O. (1 de janeiro de 2023). «Entropy and the arrow of time in population dynamics». Biosystems (em inglês). 104817 páginas. ISSN 0303-2647. doi:10.1016/j.biosystems.2022.104817. Consultado em 1 de julho de 2023
-
↑ Kondepudi, Dilip K.; De Bari, Benjamin; Dixon, James A. (novembro de 2020). «Dissipative Structures, Organisms and Evolution». Entropy (em inglês) (11). 1305 páginas. ISSN 1099-4300. doi:10.3390/e22111305. Consultado em 12 de maio de 2024
-
↑ Yockey, Hubert P. (outubro de 1995). «Comments on "Let there be Life"; Thermodynamic Reflections on Biogenesis and Evolution" by Avshalom C. Elitzur». Journal of Theoretical Biology (3): 349–355. ISSN 0022-5193. doi:10.1006/jtbi.1995.0204. Consultado em 12 de maio de 2024
-
↑ PAUL, C. R. C. (junho de 1997). <191::aid-gj725>3.0.co;2-z «Book Review: NEW APPROACHES TO SPECIATION IN THE FOSSIL RECORD. Eds. Douglas H. Erwin and Robert L. Anstey. Columbia University Press, 1995. 342 pp. £34.00 ($49.50) (hardback). ISBN 0 231 08248 7». Geological Journal (2): 191–191. ISSN 0072-1050. doi:10.1002/(sici)1099-1034(199706)32:2<191::aid-gj725>3.0.co;2-z. Consultado em 23 de julho de 2023
-
1 2 Rabosky, Daniel L.; Lovette, Irby J. (agosto de 2008). «Explosive evolutionary radiations: decreasing speciation or increasing extinction through time?». Evolution; International Journal of Organic Evolution (8): 1866–1875. ISSN 0014-3820. PMID 18452577. doi:10.1111/j.1558-5646.2008.00409.x. Consultado em 6 de dezembro de 2022
-
1 2 Neto, Sodré; Fernandes Alves, Everton (12 de maio de 2017). «Speciation in real time and historical-archaeological and its absence in geological time». web.archive.org. Academia Journal of Scientific Research. Consultado em 24 de outubro de 2020
-
↑ Жахов, Николай Владимирович (22 de julho de 2020). «МИРОВОЗЗРЕНЧЕСКИЕ АСПЕКТЫ МЕТОДОЛОГИИ ГОСУДАРСТВЕННОГО РЕГУЛИРОВАНИЯ АГРАРНОГО СЕКТОРА ЭКОНОМИКИ РЕГИОНА». Региональные проблемы преобразования экономики (11): 35–41. ISSN 1812-7096. doi:10.26726/1812-7096-2019-11-35-41. Consultado em 23 de julho de 2023
-
↑ Vopson, Melvin M. (1 de outubro de 2023). «The second law of infodynamics and its implications for the simulated universe hypothesis». AIP Advances (10). ISSN 2158-3226. doi:10.1063/5.0173278. Consultado em 12 de maio de 2024
-
↑ Davidson, Eric H.; Rast, Jonathan P.; Oliveri, Paola; Ransick, Andrew; Calestani, Cristina; Yuh, Chiou-Hwa; Minokawa, Takuya; Amore, Gabriele; Hinman, Veronica (março de 2002). «A Genomic Regulatory Network for Development». Science (em inglês) (5560): 1669–1678. ISSN 0036-8075. doi:10.1126/science.1069883. Consultado em 24 de outubro de 2023
-
↑ Carroll, Patrick A.; Freie, Brian W.; Mathsyaraja, Haritha; Eisenman, Robert N. (1 de agosto de 2018). «The MYC transcription factor network: balancing metabolism, proliferation and oncogenesis». Frontiers of Medicine (em inglês) (4): 412–425. ISSN 2095-0225. PMC 7358075
 . PMID 30054853. doi:10.1007/s11684-018-0650-z. Consultado em 24 de outubro de 2023
. PMID 30054853. doi:10.1007/s11684-018-0650-z. Consultado em 24 de outubro de 2023 -
↑ Lim, Jormay; Thiery, Jean Paul (1 de outubro de 2012). «Epithelial-mesenchymal transitions: insights from development». Development (19): 3471–3486. ISSN 1477-9129. doi:10.1242/dev.071209. Consultado em 24 de outubro de 2023
-
↑ Thomas, B. D. (19 de outubro de 2018). «Collagen remnants in ancient bone» (em inglês). doi:10.17638/03033541. Consultado em 1 de agosto de 2023
-
↑ 120 peer-reviewed journal articles on surviving endogenous biological material including tissue and DNA. «List of Biomaterial Fossil Papers». Google Docs. Consultado em 1 de agosto de 2023
-
↑ Yeoman, Barry. «Schweitzer's Dangerous Discovery». Discover Magazine (em inglês). Consultado em 1 de agosto de 2023
-
↑ Ehrlich, H.; Rigby, J. Keith; Botting, J. P.; Tsurkan, M. V.; Werner, C.; Schwille, P.; Petrášek, Z.; Pisera, A.; Simon, P. (13 de dezembro de 2013). «Discovery of 505-million-year old chitin in the basal demosponge Vauxia gracilenta». Scientific Reports (em inglês) (1). 3497 páginas. ISSN 2045-2322. doi:10.1038/srep03497. Consultado em 24 de julho de 2023
-
↑ Anderson, Landon A. (1 de maio de 2023). «A chemical framework for the preservation of fossil vertebrate cells and soft tissues». Earth-Science Reviews (em inglês). 104367 páginas. ISSN 0012-8252. doi:10.1016/j.earscirev.2023.104367. Consultado em 24 de julho de 2023
-
↑ Thomas, Brian; Taylor, Stephen (2 de dezembro de 2019). «Proteomes of the past: the pursuit of proteins in paleontology». Expert Review of Proteomics (em inglês) (11-12): 881–895. ISSN 1478-9450. doi:10.1080/14789450.2019.1700114. Consultado em 24 de julho de 2023
-
↑ «Schweitzer's Dangerous Discovery | Dinosaurs | DISCOVER Magazine». web.archive.org. 20 de outubro de 2012. Consultado em 1 de agosto de 2023
-
↑ Senter, Philip J. (1 de agosto de 2022). «Radiocarbon in Dinosaur Bones Revisited». The American Biology Teacher (em inglês) (6): 336–341. ISSN 0002-7685. doi:10.1525/abt.2022.84.6.336. Consultado em 12 de maio de 2024
-
↑ Widacki, Michał (1 de dezembro de 2017). «Tuvya Amsel, Practicing Polygraph, best practice guide, CreateSpace Independent Publishing Platform, North Charleston, 2017». European Polygraph (4): 183–186. ISSN 1898-5238. doi:10.1515/ep-2017-0018. Consultado em 11 de junho de 2023
-
↑ MAUGER, RL (1977). «K-AR AGES OF BIOTITES FROM TUFFS IN EOCENE ROCKS OF THE GREEN RIVER, WASHAKIE, AND UINTA BASINS, UTAH, WYOMING, AND COLORADO». K-AR AGES OF BIOTITES FROM TUFFS IN EOCENE ROCKS OF THE GREEN RIVER, WASHAKIE, AND UINTA BASINS, UTAH, WYOMING, AND COLORADO. Consultado em 11 de junho de 2023
-
↑ pubs.geoscienceworld.org https://pubs.geoscienceworld.org/uwyo/rmg/article-standard/15/1/17/87712/K-Ar-ages-of-biotites-from-tuffs-in-Eocene-rocks. Consultado em 11 de junho de 2023 Em falta ou vazio
|título=(ajuda) -
↑ Paul, C. R. C. (Christopher R. C. ) (1980). The natural history of fossils. Internet Archive. [S.l.]: London : Weidenfeld and Nicolson
-
↑ «Is Radiometric Dating Accurate? – Earth Age». earthage.org. Consultado em 11 de junho de 2023
-
↑ «Carbon-14 dating dinosaur bones». newgeology.us (em inglês). Consultado em 1 de agosto de 2023
-
1 2 Bergman, Jerry (2 de outubro de 2020). «Contemporary Religious Discrimination Against Creationists in Academia». Proceedings of the International Conference on Creationism (1). ISSN 2639-4006. Consultado em 6 de agosto de 2023
-
↑ Cooks, R. G.; Hao (2006). «Polar Acetalization and Transacetalization in the Gas Phase: The Eberlin Reaction». Chemical Reviews. 106 (1): 188-211. ISSN 0009-2665. doi:10.1021/cr0400921 A referência emprega parâmetros obsoletos
|coautor=(ajuda) -
↑ Zheng, Xubin; W. Andy (2001). «Eberlin reaction of arenesulfenylium cations with cyclic acetals and ketals: ring contraction and cycloreversion». Journal of the Chemical Society, Perkin Transactions 2 (3). doi:10.1039/b007941n A referência emprega parâmetros obsoletos
|coautor=(ajuda) -
↑ «Scopus preview - Eberlin, M. N. - Author details - Scopus». www.scopus.com. Consultado em 6 de agosto de 2023
-
↑ «Marcos N. Eberlin». scholar.google.com. Consultado em 6 de agosto de 2023
-
↑ «ASMS Members Honored at IMSF 2016». asms.org. Consultado em 11 de junho de 2020
-
↑ «Medalha J. J. Thomson | Instituto de Química». iqm.unicamp.br. Consultado em 11 de junho de 2020
-
↑ Eberlin, M.N. «Publicações de Marcos Nogueira Eberlin». buscatextual.cnpq.br. Consultado em 6 de agosto de 2023
-
↑ «Scopus preview - McLuckey, Scott A. - Author details - Scopus». www.scopus.com. Consultado em 6 de agosto de 2023
-
↑ «Thomson Medal and Prize» (em inglês). Consultado em 5 de maio de 2010
-
↑ Hoffmann, Edmond de (2005). «Mass Spectrometry». American Cancer Society (em inglês). ISBN 978-0-471-23896-6. doi:10.1002/0471238961.1301191913151518.a01.pub2. Consultado em 2 de fevereiro de 2021
-
↑ Haddad, Renato; Sparrapan, Regina; Eberlin, Marcos N. (2006). «Desorption sonic spray ionization for (high) voltage-free ambient mass spectrometry». Rapid Communications in Mass Spectrometry (em inglês) (19): 2901–2905. ISSN 1097-0231. doi:10.1002/rcm.2680. Consultado em 2 de fevereiro de 2021
-
↑ Ekinci, K. L.; Roukes, M. L. (26 de maio de 2005). «Nanoelectromechanical systems». Review of Scientific Instruments (6). 061101 páginas. ISSN 0034-6748. doi:10.1063/1.1927327. Consultado em 2 de fevereiro de 2021
-
↑ Eberlin, Livia. «Doutoranda brasileira ganha prêmio de química inédito nos Estados Unidos». www.unicamp.br. Consultado em 6 de agosto de 2023
-
↑ R7.com (4 de junho de 2014). «Doutoranda brasileira ganha prêmio de química inédito nos Estados Unidos». R7.com. Consultado em 6 de agosto de 2023
-
↑ Segelken, Roger (14 de maio de 1987). «Biologist invent gun for shooting cells with DNA» (PDF). Cornell Chronicle. p. 3. Consultado em 24 de abril de 2016
-
↑ Sanford, J.C.; T, .M.; Wolf, E.D.; Allen, N (1987). «Delivery of substances into cells and tissues using a particle bombardment process». Particulate Science and Technology. 5 (1): 27–37. doi:10.1080/02726358708904533
-
↑ Klein, T.M.; Wolf, E.D.; Wu, R.; Sanford, J.C. (Maio de 1987). «High-velocity microprojectiles for delivering nucleic acids into living cells». Nature. 327: 70–73. doi:10.1038/327070a0
-
↑ «Gene gun». Wikipedia (em inglês). 28 de março de 2021. Consultado em 12 de abril de 2021
-
↑ Brewer, Wesley H.; Baumgardner, John R.; Sanford, John C. (10 de abril de 2013). «Using Numerical Simulation to Test the ?Mutation-Count? Hypothesis». WORLD SCIENTIFIC: 298–311. ISBN 978-981-4508-71-1. doi:10.1142/9789814508728_0012. Consultado em 5 de abril de 2021
-
↑ Crevecoeur, Guibert U. (1 de dezembro de 2019). «Entropy growth and information gain in operating organized systems». AIP Advances (12). 125041 páginas. doi:10.1063/1.5128315. Consultado em 5 de abril de 2021
-
↑ Sodre Neto SG Neto (2021). «A TEORIA DA SOBREVIVÊNCIA DAS ESPÉCIES SOB EMPOBRECIMENTO GENÉTICO (ENTROPIA GENÉTICA) ATUANDO PELOS MOTORES EVOLUTIVOS». doi:10.13140/RG.2.2.14369.43367. Consultado em 5 de abril de 2021
-
↑ Courtois, Gilles; Morgan, John G.; Campbell, Linda A.; Fourel, Genevieve; Crabtree, Gerald R. (30 de outubro de 1987). «Interaction of a Liver-Specific Nuclear Factor with the Fibrinogen and Antitrypsin Promoters». Science (4827): 688–692. ISSN 0036-8075. doi:10.1126/science.3499668. Consultado em 24 de julho de 2023
-
↑ Crabtree, Gerald R.; Kant, Jeffrey A. (novembro de 1982). «Organization of the rat γ-fibrinogen gene: Alternative mRNA splice patterns produce the γA and γB (γ′) chains of fibrinogen». Cell (1): 159–166. ISSN 0092-8674. doi:10.1016/0092-8674(82)90415-9. Consultado em 24 de julho de 2023
-
↑ Dieterich, Veit-Jakobus (1 de dezembro de 2009). «„Evolution", von Reinhard Junker und Siegfried Scherer». Zeitschrift für Pädagogik und Theologie (4): 340–346. ISSN 2366-7796. doi:10.1515/zpt-2009-0406. Consultado em 23 de julho de 2023
-
↑ Sanders, Roger; Wise, Kurt (6 de outubro de 2020). «The Cognitum: A Perception-Dependent Concept Needed in Baraminology». Proceedings of the International Conference on Creationism (1). ISSN 2639-4006. Consultado em 30 de julho de 2023
-
↑ Wood, Todd Charles; Tomkins, Jeffrey P. (15 de setembro de 2006). «Genomic Sequencing (Core Article)». Weinheim, Germany: Wiley-VCH Verlag GmbH & Co. KGaA. Encyclopedia of Molecular Cell Biology and Molecular Medicine. Consultado em 30 de julho de 2023
-
↑ McLain, Matthew; Petrone, Matt; Speights, Matthew (2018). «Feathered dinosaurs reconsidered: New insights from baraminology and ethnotaxonomy». The Proceedings of the International Conference on Creationism (1): 472–515. ISSN 2639-4006. doi:10.15385/jpicc.2018.8.1.37. Consultado em 30 de julho de 2023
-
↑ «refutação do artigo». portaltdibrasil.comunidades.net. Consultado em 30 de julho de 2023
-
↑ Colombo, Matteo; Wright, Cory (1 de junho de 2021). «First principles in the life sciences: the free-energy principle, organicism, and mechanism». Synthese (em inglês) (14): 3463–3488. ISSN 1573-0964. doi:10.1007/s11229-018-01932-w. Consultado em 1 de julho de 2023
-
1 2 Henn, Ronaldo Cesar; Oliveria, Felipe Moura de (15 de julho de 2015). «Jornalismo e movimentos em rede: a emergência de uma crise sistêmica». Revista FAMECOS (3): 77–95. ISSN 1980-3729. doi:10.15448/1980-3729.2015.3.20560. Consultado em 30 de junho de 2023
-
↑ Love, Askell; Mayr, Ernst (1971). «Populations, Species, and Evolution. An Abridgment of Animal Species and Evolution». The Bryologist (2). 226 páginas. ISSN 0007-2745. doi:10.2307/3241850. Consultado em 24 de outubro de 2020
-
↑ Axe, Douglas D. (15 de abril de 2010). «The Limits of Complex Adaptation: An Analysis Based on a Simple Model of Structured Bacterial Populations». BIO-Complexity (4). ISSN 2151-7444. doi:10.5048/bio-c.2010.4. Consultado em 24 de outubro de 2020
-
↑ Charlesworth, B.; Harvey, P. H.; Charlesworth, Brian; Charlesworth, Deborah (29 de novembro de 2000). «The degeneration of Y chromosomes». Philosophical Transactions of the Royal Society of London. Series B: Biological Sciences (1403): 1563–1572. PMC 1692900
 . PMID 11127901. doi:10.1098/rstb.2000.0717. Consultado em 11 de outubro de 2022
. PMID 11127901. doi:10.1098/rstb.2000.0717. Consultado em 11 de outubro de 2022 -
↑ Basener, William F.; Sanford, John C. (1 de junho de 2018). «The fundamental theorem of natural selection with mutations». Journal of Mathematical Biology (em inglês) (7): 1589–1622. ISSN 1432-1416. doi:10.1007/s00285-017-1190-x. Consultado em 16 de outubro de 2022
-
↑ Loverdo, Claude; Lloyd-Smith, James O. (17 de julho de 2013). «Evolutionary Invasion and Escape in the Presence of Deleterious Mutations». PLOS ONE (em inglês) (7): e68179. ISSN 1932-6203. PMC 3714272
 . PMID 23874532. doi:10.1371/journal.pone.0068179. Consultado em 16 de outubro de 2022
. PMID 23874532. doi:10.1371/journal.pone.0068179. Consultado em 16 de outubro de 2022 -
↑ Thorvaldsen, Steinar; Hössjer, Ola (21 de setembro de 2020). «Using statistical methods to model the fine-tuning of molecular machines and systems». Journal of Theoretical Biology (em inglês). 110352 páginas. ISSN 0022-5193. doi:10.1016/j.jtbi.2020.110352. Consultado em 16 de outubro de 2022
-
↑ Fry, James D (1 de junho de 2004). «How Common Are Overdominant Mutations?». Genetics (2): 1031–1032. ISSN 1943-2631. PMC 1470920
 . PMID 15238551. doi:10.1534/genetics.103.024471. Consultado em 16 de outubro de 2022
. PMID 15238551. doi:10.1534/genetics.103.024471. Consultado em 16 de outubro de 2022 -
↑ Gillespie, J H (1994). «Substitution processes in molecular evolution. III. Deleterious alleles». academic.oup.com. PMC 1206239
 . PMID 7851786. doi:10.1093/genetics/138.3.943. Consultado em 16 de outubro de 2022
. PMID 7851786. doi:10.1093/genetics/138.3.943. Consultado em 16 de outubro de 2022 -
↑ Sniegowski, Paul D.; Gerrish, Philip J.; Lenski, Richard E. (junho de 1997). «Evolution of high mutation rates in experimental populations of E. coli». Nature (em inglês) (6634): 703–705. ISSN 1476-4687. doi:10.1038/42701. Consultado em 16 de outubro de 2022
-
↑ Woodruff, Ronny C.; Balinski, Michael A.; Bouzat, Juan L. (outubro de 2015). «A perspective on the evolution of germ-cell development and germinal mosaics of deleterious mutations». Genetica (5): 563–569. ISSN 1573-6857. PMID 26113303. doi:10.1007/s10709-015-9854-1. Consultado em 16 de outubro de 2022
-
↑ Bataillon, Thomas (maio de 2000). «Estimation of spontaneous genome-wide mutation rate parameters: whither beneficial mutations?». Heredity (em inglês) (5): 497–501. ISSN 1365-2540. doi:10.1046/j.1365-2540.2000.00727.x. Consultado em 17 de dezembro de 2022
-
↑ Gerrish, Philip J.; Lenski, Richard E. (1 de março de 1998). «The fate of competing beneficial mutations in an asexual population». Genetica (em inglês) (0). 127 páginas. ISSN 1573-6857. doi:10.1023/A:1017067816551. Consultado em 11 de abril de 2022
-
↑ Basener, William F.; Sanford, John C. (1 de junho de 2018). «The fundamental theorem of natural selection with mutations». Journal of Mathematical Biology (em inglês). 76 (7): 1589–1622. ISSN 1432-1416. PMC 5906570
 . PMID 29116373. doi:10.1007/s00285-017-1190-x
. PMID 29116373. doi:10.1007/s00285-017-1190-x -
↑ Nelson, Chase W.; Sanford, John C. (10 de abril de 2013). «Computational Evolution Experiments Reveal a Net Loss of Genetic Information Despite Selection». WORLD SCIENTIFIC: 338–368. ISBN 9789814508711. doi:10.1142/9789814508728_0014
-
↑ Bürger, Reinhard (1989). Kurzhanski, A. B.; Sigmund, K., eds. «Mutation-Selection Models in Population Genetics and Evolutionary Game Theory». Springer Netherlands. Evolution and Control in Biological Systems (em inglês): 75–89. ISBN 9789400923584. doi:10.1007/978-94-009-2358-4_8
-
↑ Carter, Robert W.; Sanford, John C. (12 de outubro de 2012). «A new look at an old virus: patterns of mutation accumulation in the human H1N1 influenza virus since 1918». Theoretical Biology and Medical Modelling. 9 (1). 42 páginas. ISSN 1742-4682. PMC 3507676
 . PMID 23062055. doi:10.1186/1742-4682-9-42
. PMID 23062055. doi:10.1186/1742-4682-9-42 -
↑ Aris-Brosou, Stéphane (25 de abril de 2018). «Direct evidence of an increasing mutational load in humans». dx.doi.org (publicado em 2019). pp. 2823–2829. doi:10.1093/molbev/msz192. Consultado em 17 de dezembro de 2022 – via Molecular Biology and Evolution
-
↑ Montañez, George; Marks II, Robert J.; Fernandez, Jorge; Sanford, John C. (10 de abril de 2013). «Multiple Overlapping Genetic Codes Profoundly Reduce the Probability of Beneficial Mutation». WORLD SCIENTIFIC: 139–167. ISBN 9789814508711. doi:10.1142/9789814508728_0006
-
↑ Brewer, Wesley H.; Baumgardner, John R.; Sanford, John C. (10 de abril de 2013). «Using Numerical Simulation to Test the ?Mutation-Count? Hypothesis». WORLD SCIENTIFIC: 298–311. ISBN 9789814508711. doi:10.1142/9789814508728_0012
-
↑ Thornton, Kevin R.; Long, Anthony D.; Sanjak, Jaleal S. (19 de janeiro de 2017). «A Model of Compound Heterozygous, Loss-of-Function Alleles Is Broadly Consistent with Observations from Complex-Disease GWAS Datasets». PLOS Genetics (em inglês). 13 (1): e1006573. ISSN 1553-7404. doi:10.1371/journal.pgen.1006573
-
↑ Lynch, Michael (19 de janeiro de 2010). «Rate, molecular spectrum, and consequences of human mutation». Proceedings of the National Academy of Sciences (em inglês) (3): 961–968. ISSN 0027-8424. PMC 2824313
 . PMID 20080596. doi:10.1073/pnas.0912629107. Consultado em 11 de outubro de 2022
. PMID 20080596. doi:10.1073/pnas.0912629107. Consultado em 11 de outubro de 2022 -
↑ Patel, Ravi K.; Jain, Mukesh (2012). «NGS QC Toolkit: A Toolkit for Quality Control of Next Generation Sequencing Data». PLOS ONE (em inglês). 7 (2): e30619. ISSN 1932-6203. PMC 3270013
 . PMID 22312429. doi:10.1371/journal.pone.0030619
. PMID 22312429. doi:10.1371/journal.pone.0030619 -
↑ Zeeshan, Saman; Xiong, Ruoyun; Liang, Bruce T.; Ahmed, Zeeshan (18 de maio de 2020). «100 Years of evolving gene–disease complexities and scientific debutants». Briefings in Bioinformatics (em inglês). 21 (3): 885–905. doi:10.1093/bib/bbz038
-
↑ Oliveira, Daniela; Miranda, Filipe Manuel Mota; Coimbra, Ana; Abreu, Nuno; Leuschner, Pedro; Abelha, António Carlos (1 de janeiro de 2020). «OpenEHR Meets Interoperability and Knowledge Engineering». International Journal of Reliable and Quality E-Healthcare (IJRQEH) (em inglês). Consultado em 20 de setembro de 2020
-
↑ Hak, Francini; Oliveira, Daniela; Abreu, Nuno; Leuschner, Pedro; Abelha, António; Santos, Manuel (1 de janeiro de 2020). «An OpenEHR Adoption in a Portuguese Healthcare Facility». Procedia Computer Science. The 11th International Conference on Ambient Systems, Networks and Technologies (ANT) / The 3rd International Conference on Emerging Data and Industry 4.0 (EDI40) / Affiliated Workshops (em inglês): 1047–1052. ISSN 1877-0509. doi:10.1016/j.procs.2020.03.075. Consultado em 20 de setembro de 2020
-
↑ Li, Mengyang; Leslie, Heather; Qi, Bin; Nan, Shan; Feng, Hongshuo; Cai, Hailing; Lu, Xudong; Duan, Huilong (2020). «Development of an openEHR Template for COVID-19 Based on Clinical Guidelines». Journal of Medical Internet Research (em inglês) (6): e20239. doi:10.2196/20239. Consultado em 20 de setembro de 2020
-
↑ Mascia, Cecilia; Uva, Paolo; Leo, Simone; Zanetti, Gianluigi (28 de setembro de 2017). «OpenEHR modeling for genomics in clinical practice». dx.doi.org. Consultado em 20 de setembro de 2020
-
↑ Nørregaard Jensen, Ole (1 de fevereiro de 2004). «Modification-specific proteomics: characterization of post-translational modifications by mass spectrometry». Current Opinion in Chemical Biology (em inglês). 8 (1): 33–41. ISSN 1367-5931. doi:10.1016/j.cbpa.2003.12.009
-
↑ Gligorijević, Vladimir; Malod‐Dognin, Noël; Pržulj, Nataša (2016). «Integrative methods for analyzing big data in precision medicine». PROTEOMICS (em inglês). 16 (5): 741–758. ISSN 1615-9861. doi:10.1002/pmic.201500396
-
↑ Zaidi, Shabi Abbas; Shahzad, Faisal; Batool, Sadaf (2020). «Progress in cancer biomarkers monitoring strategies using graphene modified support materials». Talanta. 210. 120669 páginas. ISSN 0039-9140. doi:10.1016/j.talanta.2019.120669
-
↑ Tyanova, Stefka; Temu, Tikira; Sinitcyn, Pavel; Carlson, Arthur; Hein, Marco Y.; Geiger, Tamar; Mann, Matthias; Cox, Jürgen (2016). «The Perseus computational platform for comprehensive analysis of (prote)omics data». Nature Methods (em inglês). 13 (9): 731–740. ISSN 1548-7105. doi:10.1038/nmeth.3901
-
↑ Perez-Riverol, Yasset; Csordas, Attila; Bai, Jingwen; Bernal-Llinares, Manuel; Hewapathirana, Suresh; Kundu, Deepti J.; Inuganti, Avinash; Griss, Johannes; Mayer, Gerhard (8 de janeiro de 2019). «The PRIDE database and related tools and resources in 2019: improving support for quantification data». Nucleic Acids Research (em inglês). 47 (D1): D442–D450. ISSN 0305-1048. doi:10.1093/nar/gky1106
-
↑ Biswas, Nupur; Kumar, Krishna; Bose, Sarpita; Bera, Raisa; Chakrabarti, Saikat (20 de abril de 2020). «Analysis of Pan-Omics Data in Human Interactome Network (APODHIN)». bioRxiv (em inglês): 2020.04.18.048207. doi:10.1101/2020.04.18.048207
-
↑ Fu, Wenqing; O'Connor, Timothy D.; Jun, Goo; Kang, Hyun Min; Abecasis, Goncalo; Leal, Suzanne M.; Gabriel, Stacey; Rieder, Mark J.; Altshuler, David (10 de janeiro de 2013). «Analysis of 6,515 exomes reveals the recent origin of most human protein-coding variants». Nature. 493 (7431): 216–220. ISSN 1476-4687. PMC 3676746
 . PMID 23201682. doi:10.1038/nature11690
. PMID 23201682. doi:10.1038/nature11690 -
↑ Saey, Tina Hesman (14 de agosto de 2012). «Uncommon carriers: People have a surprising number of rare genetic variants». Science News. 182 (4): 28–29. ISSN 0036-8423. doi:10.1002/scin.5591820421
-
↑ Consortium, Exome Aggregation; MacArthur, Daniel G.; Daly, Mark J.; Wilson, James G.; Watkins, Hugh C.; Tsuang, Ming T.; Tuomilehto, Jaakko; Sullivan, Patrick F.; Sklar, Pamela (1 de agosto de 2016). «Analysis of protein-coding genetic variation in 60,706 humans». Nature (em inglês). 536 (7616): 285–291. ISSN 1476-4687. doi:10.1038/nature19057
-
1 2 Caldecott, Keith W.; Ward, Michael E.; Nussenzweig, André (fevereiro de 2022). «The threat of programmed DNA damage to neuronal genome integrity and plasticity». Nature Genetics (2): 115–120. ISSN 1546-1718. doi:10.1038/s41588-021-01001-y. Consultado em 11 de abril de 2022
-
1 2 3 Lodato, Michael A.; Rodin, Rachel E.; Bohrson, Craig L.; Coulter, Michael E.; Barton, Alison R.; Kwon, Minseok; Sherman, Maxwell A.; Vitzthum, Carl M.; Luquette, Lovelace J. (2 de fevereiro de 2018). «Aging and neurodegeneration are associated with increased mutations in single human neurons». Science (New York, N.Y.) (6375): 555–559. ISSN 1095-9203. PMC 5831169
 . PMID 29217584. doi:10.1126/science.aao4426. Consultado em 11 de abril de 2022
. PMID 29217584. doi:10.1126/science.aao4426. Consultado em 11 de abril de 2022 -
↑ Monday, Hannah R.; Younts, Thomas J.; Castillo, Pablo E. (2018). «Long-Term Plasticity of Neurotransmitter Release: Emerging Mechanisms and Contributions to Brain Function and Disease». Annual Review of Neuroscience. 41 (1): 299–322. PMID 29709205. doi:10.1146/annurev-neuro-080317-062155
-
↑ Sullivan, Patrick F.; Geschwind, Daniel H. (21 de março de 2019). «Defining the Genetic, Genomic, Cellular, and Diagnostic Architectures of Psychiatric Disorders». Cell. 177 (1): 162–183. ISSN 0092-8674. doi:10.1016/j.cell.2019.01.015
-
↑ Risch, Neil; Hoffmann, Thomas J.; Anderson, Meredith; Croen, Lisa A.; Grether, Judith K.; Windham, Gayle C. (1 de novembro de 2014). «Familial Recurrence of Autism Spectrum Disorder: Evaluating Genetic and Environmental Contributions». American Journal of Psychiatry. 171 (11): 1206–1213. ISSN 0002-953X. doi:10.1176/appi.ajp.2014.13101359
-
1 2 Hickman, Richard A.; O'Shea, Sarah A.; Mehler, Mark F.; Chung, Wendy K. (fevereiro de 2022). «Neurogenetic disorders across the lifespan: from aberrant development to degeneration». Nature Reviews. Neurology (2): 117–124. ISSN 1759-4766. doi:10.1038/s41582-021-00595-5. Consultado em 11 de abril de 2022
-
↑ CHARGE; Boehnke, M.; McCarthy, M. I.; Florez, J. C.; Morris, A. P.; Scott, L. J.; Burtt, N. P.; Altshuler, D.; Rotter, J. I. (2019). «Exome sequencing of 20,791 cases of type 2 diabetes and 24,440 controls». Nature (em inglês). 570. ISSN 0028-0836
-
↑ Vaxillaire, Martine; Froguel, Philippe; Bonnefond, Amélie (5 de agosto de 2019). «How Recent Advances in Genomics Improve Precision Diagnosis and Personalized Care of Maturity-Onset Diabetes of the Young». Current Diabetes Reports (em inglês). 19 (9). 79 páginas. ISSN 1539-0829. doi:10.1007/s11892-019-1202-x
-
↑ Morales, Ana; Hedges, Dale J.; Hershberger, Ray E. (1 de setembro de 2013). «Dilated cardiomyopathy: the complexity of a diverse genetic architecture». Nature Reviews Cardiology (em inglês). 10 (9): 531–547. ISSN 1759-5010. doi:10.1038/nrcardio.2013.105
-
↑ Borghi-Silva, Audrey; Arena, Ross; Bonjorno Júnior, José C.; Caruso, Flávia C. R.; Arakelian, Vivian M.; Tossini, Natália; Mendes, Renata G.; Cabiddu, Ramona; Bassi, Daniela (1 de julho de 2018). «Effects of Coexistence Hypertension and Type II Diabetes on Heart Rate Variability and Cardiorespiratory Fitness». Arquivos Brasileiros de Cardiologia (em inglês). 111 (1): 64–72. ISSN 0066-782X. doi:10.5935/abc.20180105
-
↑ Carey, David J.; Baras, Aris; Ledbetter, David H.; Reid, Jeffrey G.; Shuldiner, Alan R.; Ritchie, Marylyn D.; Gottesman, Omri; Faucett, William A.; Davis, F. Daniel (23 de dezembro de 2016). «Distribution and clinical impact of functional variants in 50,726 whole-exome sequences from the DiscovEHR study». Science (em inglês). 354 (6319): aaf6814. ISSN 0036-8075. PMID 28008009. doi:10.1126/science.aaf6814
-
↑ Basener, William F.; Sanford, John C. (1 de junho de 2018). «The fundamental theorem of natural selection with mutations». Journal of Mathematical Biology (em inglês). 76 (7): 1589–1622. ISSN 1432-1416. PMC 5906570
 . PMID 29116373. doi:10.1007/s00285-017-1190-x
. PMID 29116373. doi:10.1007/s00285-017-1190-x -
↑ Baumgardner, John R.; Brewer, Wesley H.; Sanford, John C. (10 de abril de 2013). «Can Synergistic Epistasis Halt Mutation Accumulation? Results from Numerical Simulation». WORLD SCIENTIFIC: 312–337. ISBN 9789814508711. doi:10.1142/9789814508728_0013
-
↑ Campbell, Catarina D.; Chong, Jessica X.; Malig, Maika; Ko, Arthur; Dumont, Beth L.; Han, Lide; Vives, Laura; O'Roak, Brian J.; Sudmant, Peter H. (novembro de 2012). «Estimating the human mutation rate using autozygosity in a founder population». Nature Genetics (em inglês) (11): 1277–1281. ISSN 1546-1718. doi:10.1038/ng.2418. Consultado em 24 de julho de 2023
-
↑ Modabbernia, Amirhossein; Ashrafi, Mandana; Modabbernia, Mohammad Jafar (agosto de 2010). «Lets try erythropoietin in Alzheimer's disease». Medical Hypotheses (2): 270–271. ISSN 0306-9877. doi:10.1016/j.mehy.2010.03.025. Consultado em 24 de julho de 2023
-
↑ Konermann, Silvana; Lotfy, Peter; Brideau, Nicholas J.; Oki, Jennifer; Shokhirev, Maxim N.; Hsu, Patrick D. (abril de 2018). «Transcriptome Engineering with RNA-Targeting Type VI-D CRISPR Effectors». Cell (3): 665–676.e14. ISSN 0092-8674. doi:10.1016/j.cell.2018.02.033. Consultado em 24 de julho de 2023
-
↑ Doudna, Jennifer A.; Charpentier, Emmanuelle (28 de novembro de 2014). «The new frontier of genome engineering with CRISPR-Cas9». Science (6213). ISSN 0036-8075. doi:10.1126/science.1258096. Consultado em 24 de julho de 2023
-
↑ Erkut, Esra; Yokota, Toshifumi (18 de janeiro de 2022). «CRISPR Therapeutics for Duchenne Muscular Dystrophy». dx.doi.org. Consultado em 24 de julho de 2023
-
↑ Riepe, C.; Kopito, R. (novembro de 2021). «640: Genome-wide CRISPR/Cas9 analyses of F508del-CFTR degradation». Journal of Cystic Fibrosis: S304. ISSN 1569-1993. doi:10.1016/s1569-1993(21)02063-4. Consultado em 24 de julho de 2023
-
↑ Shoop, Wendy K.; Bacman, Sandra R.; Barrera-Paez, Jose Domingo; Moraes, Carlos T. (16 de março de 2023). «Mitochondrial gene editing». Nature Reviews Methods Primers (em inglês) (1): 1–18. ISSN 2662-8449. doi:10.1038/s43586-023-00200-7. Consultado em 30 de julho de 2023
-
↑ Al-Ibraheemi, Zainab; Ashmead, Graham; Porat, Natalie; Taylor, Dyese; Sinha, Anila (maio de 2017). «Zika Screening Beyond Brazil: Zika Madness and Cost of Reassurance? [15M]». Obstetrics & Gynecology (1): S136–S136. ISSN 0029-7844. doi:10.1097/01.aog.0000514686.29040.4b. Consultado em 24 de julho de 2023
-
↑ Arslan, Ruben C.; Willführ, Kai P.; Frans, Emma M.; Verweij, Karin J. H.; Bürkner, Paul-Christian; Myrskylä, Mikko; Voland, Eckart; Almqvist, Catarina; Zietsch, Brendan P. (13 de setembro de 2017). «Older fathers' children have lower evolutionary fitness across four centuries and in four populations». Proceedings of the Royal Society B: Biological Sciences (1862). 20171562 páginas. PMC 5597845
 . PMID 28904145. doi:10.1098/rspb.2017.1562. Consultado em 17 de dezembro de 2022
. PMID 28904145. doi:10.1098/rspb.2017.1562. Consultado em 17 de dezembro de 2022 -
↑ Bataillon, Thomas (maio de 2000). «Estimation of spontaneous genome-wide mutation rate parameters: whither beneficial mutations?». Heredity (em inglês) (5): 497–501. ISSN 1365-2540. doi:10.1046/j.1365-2540.2000.00727.x. Consultado em 17 de dezembro de 2022
-
↑ Smith, Thomas; Ho, Gladys; Christodoulou, John; Price, Elizabeth Ann; Onadim, Zerrin; Gauthier-Villars, Marion; Dehainault, Catherine; Houdayer, Claude; Parfait, Beatrice (maio de 2016). «Extensive Variation in the Mutation Rate Between and Within Human Genes Associated with Mendelian Disease: HUMAN MUTATION». Human Mutation (em inglês) (5): 488–494. doi:10.1002/humu.22967. Consultado em 17 de dezembro de 2022
-
↑ Crabtree, Gerald R. (1 de janeiro de 2013). «Our fragile intellect. Part I». Trends in Genetics (em inglês). 29 (1): 1–3. ISSN 0168-9525. doi:10.1016/j.tig.2012.10.002
-
↑ Lodato, Michael A.; Rodin, Rachel E.; Bohrson, Craig L.; Coulter, Michael E.; Barton, Alison R.; Kwon, Minseok; Sherman, Maxwell A.; Vitzthum, Carl M.; Luquette, Lovelace J. (2 de fevereiro de 2018). «Aging and neurodegeneration are associated with increased mutations in single human neurons». Science (em inglês). 359 (6375): 555–559. ISSN 0036-8075. PMID 29217584. doi:10.1126/science.aao4426
-
↑ Lynch, Michael (1 de março de 2016). «Mutation and Human Exceptionalism: Our Future Genetic Load». Genetics (em inglês). 202 (3): 869–875. ISSN 0016-6731. PMID 26953265. doi:10.1534/genetics.115.180471
-
↑ Crabtree, Gerald R. (janeiro de 2013). «Our fragile intellect. Part II». Trends in genetics: TIG (1): 3–5. ISSN 0168-9525. PMID 23153597. doi:10.1016/j.tig.2012.10.003. Consultado em 11 de abril de 2022
-
↑ Fu, Wenqing; O'Connor, Timothy D.; Jun, Goo; Kang, Hyun Min; Abecasis, Goncalo; Leal, Suzanne M.; Gabriel, Stacey; Rieder, Mark J.; Altshuler, David (10 de janeiro de 2013). «Analysis of 6,515 exomes reveals the recent origin of most human protein-coding variants». Nature (7431): 216–220. ISSN 1476-4687. PMC 3676746
 . PMID 23201682. doi:10.1038/nature11690. Consultado em 24 de outubro de 2020
. PMID 23201682. doi:10.1038/nature11690. Consultado em 24 de outubro de 2020 -
↑ «BLAST: Basic Local Alignment Search Tool». blast.ncbi.nlm.nih.gov (em inglês). Consultado em 11 de abril de 2022
-
↑ Stenson, Peter D.; Mort, Matthew; Ball, Edward V.; Evans, Katy; Hayden, Matthew; Heywood, Sally; Hussain, Michelle; Phillips, Andrew D.; Cooper, David N. (1 de junho de 2017). «The Human Gene Mutation Database: towards a comprehensive repository of inherited mutation data for medical research, genetic diagnosis and next-generation sequencing studies». Human Genetics (em inglês) (6): 665–677. ISSN 1432-1203. PMC 5429360
 . PMID 28349240. doi:10.1007/s00439-017-1779-6. Consultado em 11 de abril de 2022
. PMID 28349240. doi:10.1007/s00439-017-1779-6. Consultado em 11 de abril de 2022 -
↑ Sanford, John; Carter, Robert; Brewer, Wes; Baumgardner, John; Potter, Bruce; Potter, Jon (2018). «Adam and Eve, designed diversity, and allele frequencies». The Proceedings of the International Conference on Creationism (1): 200–216. ISSN 2639-4006. doi:10.15385/jpicc.2018.8.1.20. Consultado em 11 de abril de 2022
-
↑ Carter, Robert; Lee, Stephen; Sanford, John (27 de julho de 2018). «An Overview of the Independent Histories of the Human Y Chromosome and the Human Mitochondrial chromosome». Proceedings of the International Conference on Creationism (1). ISSN 2639-4006. doi:10.15385/jpicc.2018.8.1.15. Consultado em 11 de abril de 2022
-
↑ Cooper, David N.; Chen, Jian-Min; Ball, Edward V.; Howells, Katy; Mort, Matthew; Phillips, Andrew D.; Chuzhanova, Nadia; Krawczak, Michael; Kehrer-Sawatzki, Hildegard (13 de abril de 2010). «Genes, mutations, and human inherited disease at the dawn of the age of personalized genomics». Human Mutation (em inglês) (6): 631–655. doi:10.1002/humu.21260. Consultado em 11 de abril de 2022
-
↑ Belyeu, Jonathan R.; Brand, Harrison; Wang, Harold; Zhao, Xuefang; Pedersen, Brent S.; Feusier, Julie; Gupta, Meenal; Nicholas, Thomas J.; Brown, Joseph (1 de abril de 2021). «De novo structural mutation rates and gamete-of-origin biases revealed through genome sequencing of 2,396 families». The American Journal of Human Genetics (em inglês) (4): 597–607. ISSN 0002-9297. doi:10.1016/j.ajhg.2021.02.012. Consultado em 11 de abril de 2022
-
↑ author., Eberlin, Marcos, 1959-. Foresight : how the chemistry of life reveals planning and purpose. [S.l.: s.n.] OCLC 1108779199
-
↑ Britnell, Mark (18 de março de 2019). «Universal healthcare in our lifetime? All teach, all learn». Oxford University Press (em inglês): 144–152. ISBN 978-0-19-883652-0. doi:10.1093/oso/9780198836520.003.0018. Consultado em 11 de abril de 2022
-
↑ Sanford, J.; Carter, R.; Brewer, W.; Baumgardner, J.; Potter, Bruce E.; Potter, J. (2018). «Adam and Eve, Designed Diversity, and Allele Frequencies». doi:10.15385/JPICC.2018.8.1.20. Consultado em 11 de abril de 2022
-
↑ Carter, Robert; Lee, Stephen; Sanford, John (27 de julho de 2018). «An Overview of the Independent Histories of the Human Y Chromosome and the Human Mitochondrial chromosome». Proceedings of the International Conference on Creationism (1). ISSN 2639-4006. doi:10.15385/jpicc.2018.8.1.15. Consultado em 11 de abril de 2022
-
↑ Sodré, GBN (18 de julho de 2021). «Um dos maiores cientistas da atualidade decreta em apenas 1 minuto, o fim da polêmica criacionismo versus evolucionismo». Jornal da Ciência. doi:10.13140/RG.2.2.23766.73282. Consultado em 11 de abril de 2022
-
↑ Sanford, John; Carter, Robert; Brewer, Wes; Baumgardner, John; Potter, Bruce; Potter, Jon (2018). «Adam and Eve, designed diversity, and allele frequencies». The Proceedings of the International Conference on Creationism (1): 200–216. ISSN 2639-4006. doi:10.15385/jpicc.2018.8.1.20. Consultado em 11 de abril de 2022
-
↑ Carter, R. (2007). «Mitochondrial diversity within modern human populations». Nucleic acids research. doi:10.1093/nar/gkm207. Consultado em 11 de abril de 2022
-
↑ Yan, Shi; Wang, Chuan-Chao; Zheng, Hong-Xiang; Wang, Wei; Qin, Zhen-Dong; Wei, Lan-Hai; Wang, Yi; Pan, Xue-Dong; Fu, Wen-Qing (29 de agosto de 2014). «Y Chromosomes of 40% Chinese Descend from Three Neolithic Super-Grandfathers». PLOS ONE (em inglês) (8): e105691. ISSN 1932-6203. PMC 4149484
 . PMID 25170956. doi:10.1371/journal.pone.0105691. Consultado em 6 de dezembro de 2022
. PMID 25170956. doi:10.1371/journal.pone.0105691. Consultado em 6 de dezembro de 2022 -
↑ Fu, Wenqing; O’Connor, Timothy D; Akey, Joshua M (1 de dezembro de 2013). «Genetic architecture of quantitative traits and complex diseases». Current Opinion in Genetics & Development. Genetics of system biology (em inglês) (6): 678–683. ISSN 0959-437X. doi:10.1016/j.gde.2013.10.008. Consultado em 11 de abril de 2022
-
↑ NATHANIEL., JEANSON, (2022). TRACED;HUMAN DNA'S BIG SURPRISE. [S.l.]: MASTER BOOKS. OCLC 1306278031
-
↑ Wilson, James (julho de 2021). «The Genealogical Adam and Eve: The Surprising Science of Universal Ancestry». Bulletin for Biblical Research (2): 234–236. ISSN 1065-223X. doi:10.5325/bullbiblrese.31.2.0234. Consultado em 11 de abril de 2022
-
↑ JOSHUA., SWAMIDASS, S. (2021). GENEALOGICAL ADAM AND EVE : the surprising science of universal ancestry. [S.l.]: INTERVARSITY PRESS. OCLC 1253354060
-
↑ Kivisild, T.; Shen, P.; Wall, D.; Do, B.; Sung, R.; Davis, K.; Passarino, G.; Underhill, P.; Scharfe, C. (2006). «The Role of Selection in the Evolution of Human Mitochondrial Genomes». Genetics. doi:10.1534/genetics.105.043901. Consultado em 11 de abril de 2022
-
↑ Carter, R.; Lee, Stephen; Sanford, J. (2018). «An Overview of the Independent Histories of the Human Y Chromosome and the Human Mitochondrial chromosome». doi:10.15385/JPICC.2018.8.1.15. Consultado em 11 de abril de 2022
-
↑ Carter, R. (2007). «Mitochondrial diversity within modern human populations». Nucleic acids research. doi:10.1093/nar/gkm207. Consultado em 11 de abril de 2022
-
↑ Rohde, Douglas L. T.; Olson, Steve; Chang, Joseph T. (setembro de 2004). «Modelling the recent common ancestry of all living humans». Nature (em inglês) (7008): 562–566. ISSN 1476-4687. doi:10.1038/nature02842. Consultado em 11 de abril de 2022
-
↑ Sollars, Vincent; Lu, Xiangyi; Xiao, Li; Wang, Xiaoyan; Garfinkel, Mark D.; Ruden, Douglas M. (janeiro de 2003). «Evidence for an epigenetic mechanism by which Hsp90 acts as a capacitor for morphological evolution». Nature Genetics (em inglês) (1): 70–74. ISSN 1546-1718. doi:10.1038/ng1067. Consultado em 30 de julho de 2023
-
↑ Turner, Bryan M. (27 de novembro de 2009). «Epigenetic responses to environmental change and their evolutionary implications». Philosophical Transactions of the Royal Society B: Biological Sciences (em inglês) (1534): 3403–3418. ISSN 0962-8436. PMC 2781845
 . PMID 19833651. doi:10.1098/rstb.2009.0125. Consultado em 30 de julho de 2023
. PMID 19833651. doi:10.1098/rstb.2009.0125. Consultado em 30 de julho de 2023 -
↑ Kooke, Rik; Johannes, Frank; Wardenaar, René; Becker, Frank; Etcheverry, Mathilde; Colot, Vincent; Vreugdenhil, Dick; Keurentjes, Joost J.B. (1 de fevereiro de 2015). «Epigenetic Basis of Morphological Variation and Phenotypic Plasticity inArabidopsis thaliana». The Plant Cell (2): 337–348. ISSN 1532-298X. doi:10.1105/tpc.114.133025. Consultado em 30 de julho de 2023
-
↑ Vermeij, Geerat J. (15 de jan. de 2016). «Gigantism and Its Implications for the History of Life». PLOS ONE (em inglês) (1): e0146092. ISSN 1932-6203. PMC 4714876
 . PMID 26771527. doi:10.1371/journal.pone.0146092. Consultado em 30 de julho de 2023
. PMID 26771527. doi:10.1371/journal.pone.0146092. Consultado em 30 de julho de 2023 -
↑ Lamsdell, James C.; Braddy, Simon J. (23 de abril de 2010). «Cope's Rule and Romer's theory: patterns of diversity and gigantism in eurypterids and Palaeozoic vertebrates». Biology Letters (em inglês) (2): 265–269. ISSN 1744-9561. PMC 2865068
 . PMID 19828493. doi:10.1098/rsbl.2009.0700. Consultado em 30 de julho de 2023
. PMID 19828493. doi:10.1098/rsbl.2009.0700. Consultado em 30 de julho de 2023 -
↑ Braddy, Simon J; Poschmann, Markus; Tetlie, O. Erik (23 de fevereiro de 2008). «Giant claw reveals the largest ever arthropod». Biology Letters (em inglês) (1): 106–109. ISSN 1744-9561. PMC 2412931
 . PMID 18029297. doi:10.1098/rsbl.2007.0491. Consultado em 30 de julho de 2023
. PMID 18029297. doi:10.1098/rsbl.2007.0491. Consultado em 30 de julho de 2023 -
↑ Schoenemann, Brigitte; Poschmann, Markus; Clarkson, Euan N. K. (28 de novembro de 2019). «Insights into the 400 million-year-old eyes of giant sea scorpions (Eurypterida) suggest the structure of Palaeozoic compound eyes». Scientific Reports (em inglês) (1). 17797 páginas. ISSN 2045-2322. doi:10.1038/s41598-019-53590-8. Consultado em 30 de julho de 2023
-
↑ Briggs, Derek E.G.; Roach, Brian T. (janeiro de 2020). «Excavating eurypterids, giant arthropods of the Palaeozoic». Geology Today (em inglês) (1): 16–21. ISSN 0266-6979. doi:10.1111/gto.12296. Consultado em 30 de julho de 2023
-
↑ Elvis, A. M.; Ekta, J. S. (2011). «Ozone therapy: A clinical review». Journal of Natural Science, Biology, and Medicine. 2 (1): 66–70. ISSN 0976-9668. PMC 3312702
 . PMID 22470237. doi:10.4103/0976-9668.82319
. PMID 22470237. doi:10.4103/0976-9668.82319 -
↑ Hernandez, Alberto; Vinals, Montserrat; Pablos, Asuncion; Vilas, Francisco; Papadakos, Peter J.; Wijeysundera, Duminda N.; Vives, Marc (12 de junho de 2020). «Ozone therapy for patients with SARS-COV-2 pneumonia: a single-center prospective cohort study». medRxiv (em inglês): 2020.06.03.20117994. doi:10.1101/2020.06.03.20117994
-
↑ Schwartz, Adriana; Martínez-Sánchez, Gregorio; Lucía, Alejandra Menassa de; Viana, Sergio Mejía; Mita, Constanta Alina (12 de julho de 2020). «Complementary Application of the Ozonized Saline Solution in Mild and Severe Patients with Pneumonia Covid-19: A Non-randomized Pilot Study» (em inglês)
-
↑ Braidy, Nady; Izadi, Morteza; Sureda, Antoni; Jonaidi‐Jafari, Nematollah; Banki, Abdolali; Nabavi, Seyed F.; Nabavi, Seyed M. (2018). «Therapeutic relevance of ozone therapy in degenerative diseases: Focus on diabetes and spinal pain». Journal of Cellular Physiology (em inglês). 233 (4): 2705–2714. ISSN 1097-4652. doi:10.1002/jcp.26044
-
↑ Liu, Jian; Zhang, Peng; Tian, Jing; Li, Lun; Li, Jun; Tian, Jin Hui; Yang, KeHu (2015). «Ozone therapy for treating foot ulcers in people with diabetes». Cochrane Database of Systematic Reviews (em inglês) (10). ISSN 1465-1858. doi:10.1002/14651858.CD008474.pub2
-
↑ Zhang, Jing; Guan, Meiping; Xie, Cuihua; Luo, Xiangrong; Zhang, Qian; Xue, Yaoming (24 de junho de 2014). «Increased Growth Factors Play a Role in Wound Healing Promoted by Noninvasive Oxygen-Ozone Therapy in Diabetic Patients with Foot Ulcers». Oxidative Medicine and Cellular Longevity (em inglês). Consultado em 14 de agosto de 2020
-
↑ Tang, Wen-Juan; Jiang, Long; Wang, Ying; Kuang, Ze-Min (15 de dezembro de 2017). «Ozone therapy induced sinus arrest in a hypertensive patient with chronic kidney disease». Medicine. 96 (50). ISSN 0025-7974. PMC 5815785
 . PMID 29390373. doi:10.1097/MD.0000000000009265
. PMID 29390373. doi:10.1097/MD.0000000000009265 -
↑ Iovski, M. (2000). «Doppler blood flow velocity waveforms in hypertensive pregnant women». International Journal of Gynecology & Obstetrics. 70: D119–D119. ISSN 0020-7292. doi:10.1016/s0020-7292(00)84563-4
-
↑ Wang, Xiaoqi (28 de fevereiro de 2018). «Emerging roles of ozone in skin diseases». Zhong Nan Da Xue Xue Bao. Yi Xue Ban = Journal of Central South University. Medical Sciences. 43 (2): 114–123. ISSN 1672-7347. PMID 29559592. doi:10.11817/j.issn.1672-7347.2018.02.002
-
↑ Smetham, Graham P. (25 de julho de 2013). «Dawkins' Darwinism Part I: Evolution – The Greatest Illusion on Earth and the New Quantum Platonic Paradigm». DNA Decipher Journal (em inglês) (3). ISSN 2159-046X. Consultado em 10 de abril de 2022
-
↑ Sakamoto, Wataru (27 de julho de 2009). «Faculty Opinions recommendation of Exchange of genetic material between cells in plant tissue grafts.». Faculty Opinions – Post-Publication Peer Review of the Biomedical Literature. Consultado em 10 de abril de 2022
-
↑ Williams, Brandon; Ahsan, Muhammad Umair; Frank, Margaret H (1 de fevereiro de 2021). «Getting to the root of grafting-induced traits». Current Opinion in Plant Biology. Growth and development 2021 (em inglês). 101988 páginas. ISSN 1369-5266. doi:10.1016/j.pbi.2020.101988. Consultado em 10 de abril de 2022
-
↑ Lönnig, Wolf-Ekkehard (2011). The Evolution of the Long-necked Giraffe (Giraffa Camelopardalis L.): What Do We Really Know? ; Testing the Theories of Gradualism, Macromutation, and Intelligent Design ; a Scientific Treatise (em inglês). [S.l.]: MV-Verlag
-
↑ Santos, Charles Morphy Dias dos (junho de 2008). «Os dinossauros de Hennig: sobre a importância do monofiletismo para a sistemática biológica». Scientiae Studia: 179–200. ISSN 1678-3166. doi:10.1590/S1678-31662008000200003. Consultado em 11 de abril de 2022
-
↑ Santos, Charles Morphy Dias dos (junho de 2008). «Os dinossauros de Hennig: sobre a importância do monofiletismo para a sistemática biológica». Scientiae Studia: 179–200. ISSN 1678-3166. doi:10.1590/S1678-31662008000200003. Consultado em 11 de abril de 2022
-
↑ Haber, Matthew H. (2019). «Species in the Age of Discordance». Philosophy, Theory, and Practice in Biology. ISSN 2475-3025. doi:10.3998/ptpbio.16039257.0011.021. Consultado em 11 de abril de 2022
-
↑ MICHAEL., BEHE, (2019). DARWIN DEVOLVES : why evolution has failed to explain how species progress and how science ... shows it never will. [S.l.]: HARPER ONE. OCLC 1037282746
-
↑ Behe, Michael J. (31 de maio de 2013). «Theoretical Molecular Biology: Introductory Comments». WORLD SCIENTIFIC. doi:10.1142/9789814508728_others03. Consultado em 10 de abril de 2022
-
↑ author., Behe, Michael J., 1952-. Irreducible complexity. [S.l.: s.n.] OCLC 1181110519
-
↑ «James M. Tour». scholar.google.com. Consultado em 11 de abril de 2022
-
↑ Salisbury, Annis (14 de outubro de 1916). «The Tell-Tale Fossil». Scientific American (16): 348–349. ISSN 0036-8733. doi:10.1038/scientificamerican10141916-348. Consultado em 11 de abril de 2022
-
↑ Hetherington, Alexander J.; Berry, Christopher M.; Dolan, Liam (14 de junho de 2016). «Networks of highly branched stigmarian rootlets developed on the first giant trees». Proceedings of the National Academy of Sciences (em inglês) (24): 6695–6700. ISSN 0027-8424. PMC 4914198
 . PMID 27226309. doi:10.1073/pnas.1514427113. Consultado em 11 de abril de 2022
. PMID 27226309. doi:10.1073/pnas.1514427113. Consultado em 11 de abril de 2022 -
↑ Lönnig, Wolf-Ekkehard; Saedler, Heinz (31 de dezembro de 1997). «Plant transposons: contributors to evolution?». Gene. Junk DNA: The Role and the Evolution of Non-Coding Sequences (em inglês) (1): 245–253. ISSN 0378-1119. doi:10.1016/S0378-1119(97)00397-1. Consultado em 11 de abril de 2022
-
↑ Scherer, Siegfried (21 de setembro de 1983). «Basic functional states in the evolution of light-driven cyclic electron transport». Journal of Theoretical Biology (em inglês) (2): 289–299. ISSN 0022-5193. doi:10.1016/0022-5193(83)90416-2. Consultado em 11 de abril de 2022
-
↑ Lambert, Grant R. (7 de abril de 1984). «Enzymic editing mechanisms and the origin of biological information transfer». Journal of Theoretical Biology (em inglês) (3): 387–403. ISSN 0022-5193. doi:10.1016/S0022-5193(84)80098-3. Consultado em 11 de abril de 2022
-
↑ Pollard, William G. (1 de outubro de 1984). «Rumors of transcendence in physics». American Journal of Physics: 877–881. ISSN 0002-9505. doi:10.1119/1.13901. Consultado em 11 de abril de 2022
-
↑ Kenyon, D. H. (1984). Matsuno, Koichiro; Dose, Klaus; Harada, Kaoru; Rohlfing, Duane L., eds. «A Comparison of Proteinoid and Aldocyanoin Microsystems as Models of the Primordial Protocell». Boston, MA: Springer US (em inglês): 163–188. ISBN 978-1-4684-4640-1. doi:10.1007/978-1-4684-4640-1_13. Consultado em 11 de abril de 2022
-
↑ Scherer, Siegfried (1 de dezembro de 1984). «Transmembrane electron transport and the neutral theory of evolution». Origins of life (em inglês) (1): 725–731. ISSN 1573-0875. doi:10.1007/BF00933727. Consultado em 11 de abril de 2022
-
↑ Dose, Klaus (1 de dezembro de 1988). «The Origin of Life: More Questions Than Answers». Interdisciplinary Science Reviews (4): 348–356. ISSN 0308-0188. doi:10.1179/isr.1988.13.4.348. Consultado em 11 de abril de 2022
-
↑ Axe, Douglas D (18 de agosto de 2000). «Extreme functional sensitivity to conservative amino acid changes on enzyme exteriors11Edited by J. Karn». Journal of Molecular Biology (em inglês) (3): 585–595. ISSN 0022-2836. doi:10.1006/jmbi.2000.3997. Consultado em 11 de abril de 2022
-
↑ C., Sanford, John (2008). Genetic entropy & the mystery of the genome. [S.l.]: FMS Publications. OCLC 422876474
-
↑ Wu, Haibo; Xing, Na; Meng, Kaiwen; Fu, Beibei; Xue, Weiwei; Dong, Pan; Tang, Wanyan; Xiao, Yang; Liu, Gexin (2021). «Nucleocapsid Mutations R203K/G204R Increase the Infectivity, Fitness and Virulence of SARS-CoV-2». SSRN Electronic Journal. ISSN 1556-5068. doi:10.2139/ssrn.3896432. Consultado em 11 de abril de 2022
-
↑ den Boer, P.J. (1 de junho de 1999). «Natural Selection or the Non-survival of the Non-fit». Acta Biotheoretica (em inglês) (2): 83–97. ISSN 1572-8358. doi:10.1023/A:1002053820381. Consultado em 11 de abril de 2022
-
↑ Falcon-Lang, Howard J.; Bashforth, Arden R. (1 de maio de 2004). «Pennsylvanian uplands were forested by giant cordaitalean trees». Geology (5): 417–420. ISSN 0091-7613. doi:10.1130/G20371.1. Consultado em 11 de abril de 2022
-
↑ Hetherington, Alexander J.; Berry, Christopher M.; Dolan, Liam (14 de junho de 2016). «Networks of highly branched stigmarian rootlets developed on the first giant trees». Proceedings of the National Academy of Sciences (em inglês) (24): 6695–6700. ISSN 0027-8424. PMC 4914198
 . PMID 27226309. doi:10.1073/pnas.1514427113. Consultado em 11 de abril de 2022
. PMID 27226309. doi:10.1073/pnas.1514427113. Consultado em 11 de abril de 2022 -
↑ Donovan, S. K. (1 de maio de 2013). «Giant crinoid stems from the Lower Carboniferous (Mississippian) of Clitheroe, Lancashire, UK». Proceedings of the Yorkshire Geological Society (em inglês) (3): 211–218. ISSN 0044-0604. doi:10.1144/pygs2013-328. Consultado em 11 de abril de 2022
-
↑ Sanford, John C. (1 de dezembro de 1988). «The biolistic process». Trends in Biotechnology (em inglês) (12): 299–302. ISSN 0167-7799. doi:10.1016/0167-7799(88)90023-6. Consultado em 11 de abril de 2022
-
↑ Sanford, John C. (maio de 1990). «Biolistic plant transformation». Physiologia Plantarum (em inglês) (1): 206–209. ISSN 0031-9317. doi:10.1111/j.1399-3054.1990.tb05888.x. Consultado em 11 de abril de 2022
-
↑ Armaleo, Daniele; Ye, Guang-Ning; Klein, Theodore M.; Shark, Katherine B.; Sanford, John C.; Johnston, Stephen Albert (1 de fevereiro de 1990). «Biolistic nuclear transformation of Saccharomyces cerevisiae and other fungi». Current Genetics (em inglês) (2): 97–103. ISSN 1432-0983. doi:10.1007/BF00312852. Consultado em 11 de abril de 2022
-
↑ Sanford, John C. (setembro de 2000). «The development of the biolistic process». In Vitro Cellular & Developmental Biology - Plant (5): 303–308. ISSN 1054-5476. doi:10.1007/s11627-000-0056-9. Consultado em 11 de abril de 2022
-
↑ Shark, K B; Smith, F D; Harpending, P R; Rasmussen, J L; Sanford, J C (fevereiro de 1991). «Biolistic transformation of a procaryote, Bacillus megaterium». Applied and Environmental Microbiology (em inglês) (2): 480–485. ISSN 0099-2240. PMC 182736
 . PMID 1901706. doi:10.1128/aem.57.2.480-485.1991. Consultado em 11 de abril de 2022
. PMID 1901706. doi:10.1128/aem.57.2.480-485.1991. Consultado em 11 de abril de 2022 -
↑ Johnston, Stephen A.; Riedy, Mark; De Vit, Michael J.; Sanford, J. C.; McElligott, Sandra; Williams, R. Sanders (1 de janeiro de 1991). «Biolistic transformation of animal tissue». In Vitro Cellular & Developmental Biology - Plant (em inglês) (1): 11–14. ISSN 1475-2689. doi:10.1007/BF02632055. Consultado em 11 de abril de 2022
-
↑ Wise, Kurt (21 de outubro de 2020). «Baraminology: A Young-Earth Creation Biosystematic Method». Proceedings of the International Conference on Creationism (1). ISSN 2639-4006. Consultado em 11 de abril de 2022
-
↑ Sanders, Roger; Wise, Kurt (6 de outubro de 2020). «The Cognitum: A Perception-Dependent Concept Needed in Baraminology». Proceedings of the International Conference on Creationism (1). ISSN 2639-4006. Consultado em 11 de abril de 2022
-
↑ Brophy, Timothy (1 de janeiro de 2021). «A baraminological analysis of the landfowl (Aves: Galliformes)». Faculty Publications and Presentations. Consultado em 11 de abril de 2022
-
↑ FILIPPOV, D. V.; URUTSKOEV, L. I.; RUKHADZE, A. A. (fevereiro de 2006). «EFFECTS OF ATOMIC ELECTRONS ON NUCLEAR STABILITY AND RADIOACTIVE DECAY». WORLD SCIENTIFIC. doi:10.1142/9789812774354_0066. Consultado em 12 de abril de 2022
-
↑ Cardone, Fabio; Mignani, Roberto; Petrucci, Andrea (maio de 2009). «Piezonuclear decay of thorium». Physics Letters A (22): 1956–1958. ISSN 0375-9601. doi:10.1016/j.physleta.2009.03.067. Consultado em 12 de abril de 2022
-
↑ Urutskoev, Leonid I.; Filippov, Dmitry V. (2010). «Study of the Electric Explosion of Titanium Foils in Uranium Salts». Journal of Modern Physics (04): 226–235. ISSN 2153-1196. doi:10.4236/jmp.2010.14034. Consultado em 12 de abril de 2022
-
↑ Cardone, F.; Carpinteri, A.; Lacidogna, G. (novembro de 2009). «Piezonuclear neutrons from fracturing of inert solids». Physics Letters A (45): 4158–4163. ISSN 0375-9601. doi:10.1016/j.physleta.2009.09.026. Consultado em 12 de abril de 2022
-
↑ Carpinteri, A.; Manuello, A. (18 de agosto de 2010). «Geomechanical and Geochemical Evidence of Piezonuclear Fission Reactions in the Earth's Crust». Strain: 267–281. ISSN 0039-2103. doi:10.1111/j.1475-1305.2010.00766.x. Consultado em 12 de abril de 2022
-
↑ Verfasser, Carpinteri, Alberto. An indirect evidence of piezonuclear fission reactions: Geomechanical and geochemical evolution in the Earth's crust. [S.l.: s.n.] OCLC 1188591582
-
↑ Klemm, W. R. (2 de janeiro de 2017). «Accommodating religion to modern neuroscience». Mental Health, Religion & Culture. 20 (1): 1–19. ISSN 1367-4676. doi:10.1080/13674676.2017.1313826
-
↑ Reeves, Mariclair A.; Gauger, Ann K.; Axe, Douglas D. (12 de janeiro de 2014). «Enzyme Families--Shared Evolutionary History or Shared Design? A Study of the GABA-Aminotransferase Family». BIO-Complexity (em inglês) (0). ISSN 2151-7444. doi:10.5048/BIO-C.2014.4. Consultado em 24 de outubro de 2020
-
↑ Steele, Edward J.; Al-Mufti, Shirwan; Augustyn, Kenneth A.; Chandrajith, Rohana; Coghlan, John P.; Coulson, S. G.; Ghosh, Sudipto; Gillman, Mark; Gorczynski, Reginald M. (1 de agosto de 2018). «Cause of Cambrian Explosion - Terrestrial or Cosmic?». Progress in Biophysics and Molecular Biology (em inglês). 136: 3–23. ISSN 0079-6107. doi:10.1016/j.pbiomolbio.2018.03.004
-
↑ Gonzalez, Guillermo, 1963-. The privileged planet : how our place in the cosmos is designed for discovery. Washington, D.C: [s.n.] OCLC 1129696124 !CS1 manut: Nomes múltiplos: lista de autores (link)
-
↑ Dickinson, Mark (14 de junho de 2020). «Hubble Identifies Primeval Galaxies, Uncovers New Clues to the Universe's Evolution». web.archive.org. Consultado em 19 de março de 2021
-
↑ Morrow, Ashley (2 de março de 2016). «Hubble Team Breaks Cosmic Distance Record». NASA. Consultado em 19 de março de 2021
-
↑ «hubble heritage team: Topics by Science.gov». www.science.gov. Consultado em 19 de março de 2021
-
↑ Gobat, R.; Daddi, E.; Onodera, M.; Finoguenov, A.; Renzini, A.; Arimoto, N.; Bouwens, R.; Brusa, M.; Chary, R.-R. (fevereiro de 2011). «A mature cluster with X-ray emission at z=2.07». Astronomy & Astrophysics: A133. ISSN 0004-6361. doi:10.1051/0004-6361/201016084. Consultado em 19 de março de 2021
-
↑ Basener, William F.; Sanford, John C. (1 de junho de 2018). «The fundamental theorem of natural selection with mutations». Journal of Mathematical Biology (em inglês). 76 (7): 1589–1622. ISSN 1432-1416. PMC 5906570
 . PMID 29116373. doi:10.1007/s00285-017-1190-x
. PMID 29116373. doi:10.1007/s00285-017-1190-x -
↑ McShea, Daniel W. (1 de novembro de 1998). «POSSIBLE LARGEST-SCALE TRENDS IN ORGANISMAL EVOLUTION: Eight "Live Hypotheses"». Annual Review of Ecology and Systematics (em inglês). 29 (1): 293–318. ISSN 0066-4162. doi:10.1146/annurev.ecolsys.29.1.293
-
↑ Basener, William F.; Sanford, John C. (7 de novembro de 2017). «The fundamental theorem of natural selection with mutations». Journal of Mathematical Biology (em inglês): 1–34. ISSN 0303-6812. doi:10.1007/s00285-017-1190-x
-
↑ Orr, H. Allen (2000). «Adaptation and the Cost of Complexity». Evolution (em inglês). 54 (1): 13–20. ISSN 1558-5646. doi:10.1111/j.0014-3820.2000.tb00002.x
-
↑ Burke, Molly K.; Dunham, Joseph P.; Shahrestani, Parvin; Thornton, Kevin R.; Rose, Michael R.; Long, Anthony D. (30 de setembro de 2010). «Genome-wide analysis of a long-term evolution experiment with Drosophila». Nature (em inglês). 467 (7315): 587–590. ISSN 0028-0836. doi:10.1038/nature09352
-
↑ Lacy, Robert C. (1 de agosto de 1987). «Loss of Genetic Diversity from Managed Populations: Interacting Effects of Drift, Mutation, Immigration, Selection, and Population Subdivision». Conservation Biology (em inglês). 1 (2): 143–158. ISSN 1523-1739. doi:10.1111/j.1523-1739.1987.tb00023.x
-
↑ Belloni, Fabio; Nissim, Noaz; Romanello, Vincenzo; Eliezer, Shalom (26 de outubro de 2017). «Towards a measurement of α-decay lifetime variation at high pressure: The case of Th». 5th Workshop - Plasmi, Sorgenti, Biofisica ed Applicazioni (0): 70–75. doi:10.1285/i9788883051302p70. Consultado em 24 de outubro de 2020
-
↑ Hahn, H.- P.; Bobn, H.-J.; Kim, J. I. (1 de janeiro de 1976). «Survey on the Rate Perturbation of Nuclear Decay». Radiochimica Acta (1). ISSN 2193-3405. doi:10.1524/ract.1976.23.1.23. Consultado em 24 de outubro de 2020
-
↑ «Alpha decay perturbations by atomic effects at extreme conditions». Physics Letters B (em inglês) (4-5): 372–375. 2 de março de 2009. ISSN 0370-2693. doi:10.1016/j.physletb.2009.01.056. Consultado em 24 de outubro de 2020
-
↑ Jeppesen, H. B.; Byskov-Nielsen, J.; Wright, P.; Correia, J. G.; Fraile, L. M.; Fynbo, H. O. U.; Johnston, K.; Riisager, K. (1 de abril de 2007). «Alpha-decay half-life of 221Fr in different environments». The European Physical Journal A (em inglês) (1): 31–34. ISSN 1434-601X. doi:10.1140/epja/i2007-10011-9. Consultado em 24 de outubro de 2020
-
↑ «Alpha decay perturbations by atomic effects at extreme conditions». Physics Letters B (em inglês) (4-5): 372–375. 2 de março de 2009. ISSN 0370-2693. doi:10.1016/j.physletb.2009.01.056. Consultado em 24 de outubro de 2020
-
↑ van der Reest, Aaron J.; Currie, Philip J. (1 de maio de 2020). «Preservation frequency of tissue-like structures in vertebrate remains from the upper Campanian of Alberta: Dinosaur Park Formation». Cretaceous Research (em inglês). 109. 104370 páginas. ISSN 0195-6671. doi:10.1016/j.cretres.2019.104370
-
↑ Marcio Fraiberg Machado, Everton Fernandes Alves (19 de junho de 2020). «Perspectivas atuais sobre tecidos moles não mineralizados em fósseis de dinossauros não avianos». Terrae Didática. doi:10.20396/td.v16i0.8659539. Consultado em 24 de setembro de 2010
-
↑ «Answers in Genesis» (em inglês)
-
↑ Guzzi, Rodolfo; Colombo, Teresa; Paci, Paola (2018). Bizzarri, Mariano, ed. «Inverse Problems in Systems Biology: A Critical Review». New York, NY: Springer. Methods in Molecular Biology (em inglês): 69–94. ISBN 978-1-4939-7456-6. doi:10.1007/978-1-4939-7456-6_6. Consultado em 24 de outubro de 2023
-
↑ Gutenkunst, Ryan N.; Casey, Fergal P.; Waterfall, Joshua J.; Myers, Christopher R.; Sethna, James P. (dezembro de 2007). «Extracting Falsifiable Predictions from Sloppy Models». Annals of the New York Academy of Sciences (em inglês) (1): 203–211. ISSN 0077-8923. doi:10.1196/annals.1407.003. Consultado em 24 de outubro de 2023
-
↑ Code, Murray (2007). Code, Murray, ed. «Science and the Appropriation of Reality». London: Palgrave Macmillan UK (em inglês): 1–16. ISBN 978-0-230-59704-4. doi:10.1057/9780230597044_1
-
↑ Shah, Mohd Hazim (1 de janeiro de 2007). «Historicising Rationality: The Transmission of Rationality and Science to the Malay States under British Rule». Asian Journal of Social Science (em inglês). 35 (2): 216–241. ISSN 1568-5314. doi:10.1163/156853107X203441
-
↑ NW, 1615 L. St; Washington, Suite 800; Inquiries, DC 20036 USA202-419-4300 | Main202-419-4349 | Fax202-419-4372 | Media (5 de novembro de 2009). «Public Opinion on Religion and Science in the United States» (em inglês)
-
↑ Curry, Andrew (27 de fevereiro de 2009). «Creationist Beliefs Persist in Europe». Science (em inglês). 323 (5918): 1159–1159. ISSN 0036-8075. PMID 19251601. doi:10.1126/science.323.5918.1159
-
↑ Stein, Ben (18 de abril de 2008). «Expelled: No Intelligence Allowed». Rocky Mountain Pictures. Box Office Mojo. Consultado em 14 de maio de 2020
-
↑ «Expelled: No Intelligence Allowed». Wikipédia, a enciclopédia livre. 7 de abril de 2020
-
↑ Behe, Michael J.; Snoke, David W. (1 de outubro de 2004). «Simulating evolution by gene duplication of protein features that require multiple amino acid residues». Protein Science: A Publication of the Protein Society. 13 (10): 2651–2664. ISSN 0961-8368. PMC 2286568
 . PMID 15340163. doi:10.1110/ps.04802904
. PMID 15340163. doi:10.1110/ps.04802904 -
↑ Behe, Michael J. (1 de dezembro de 2010). «Experimental evolution, loss-of-function mutations, and "the first rule of adaptive evolution"». The Quarterly Review of Biology. 85 (4): 419–445. ISSN 0033-5770. PMID 21243963
-
↑ Kuhn, Joseph A. (1 de janeiro de 2012). «Dissecting Darwinism». Proceedings (Baylor University. Medical Center). 25 (1): 41–47. ISSN 0899-8280. PMC 3246854
 . PMID 22275784
. PMID 22275784 -
↑ Sheldon, Robert B.; Hoover, Richard B. (28 de agosto de 2008). «Cosmological evolution: spatial relativity and the speed of life». 7097. doi:10.1117/12.802195.short
-
↑ Denton, Michael (1986). Evolution: A theory in crisis (PDF). [S.l.]: Adler & Adler. pp. 368 pages. ISBN 091756152X. Consultado em 18 de novembro de 2018
-
↑ Denton, Michael J.; Marshall, Craig J.; Legge, Michael (7 de dezembro de 2002). «The protein folds as platonic forms: new support for the pre-Darwinian conception of evolution by natural law». Journal of Theoretical Biology. 219 (3): 325–342. ISSN 0022-5193. PMID 12419661
-
↑ Zuñiga, Jorge G. (2018). «La evolución de los sistemas bioquímicos complejos». revistas.ucr.ac.cr (em espanhol). VOL. 57 NÚM. 147 (2018): REVISTA DE FILOSOFÍA. Consultado em 18 de novembro de 2018
-
↑ Washington., Biological Society of; Washington., Biological Society of; Washington., Biological Society of; Institution., Smithsonian; Institution., Smithsonian (2004). «Proceedings of the Biological Society of Washington.» (em inglês). v.117 (2004). ISSN 0006-324X
-
↑ Weber, Bruce H. (31 de maio de 2013). «Towards a General Biology: Emergence of Life and Information from the Perspective of Complex Systems Dynamics». WORLD SCIENTIFIC. Biological Information (em inglês). ISBN 9789814508711. doi:10.1142/9789814508728_0024
-
↑ 1950-, Marks, Robert J., II (Robert Jackson), ([2013]). Biological information--new perspectives : proceedings of a symposium held May 31, 2011 through June 3, 2011 at Cornell University. [Hackensack] New Jersey: World Scientific. ISBN 9789814508728. OCLC 847526859
-
↑ «Emergence of Life and Biological Selection from the Perspective of Complex Systems Dynamics | Request PDF». ResearchGate (em inglês). Consultado em 18 de novembro de 2018
-
↑ Grubesich, Fabio. «THE ORIGINS OF ORDER Self-Organization and Selection in Evolution» (em inglês)
-
↑ Meester, Ronald (1 de setembro de 2009). «Simulation of biological evolution and the NFL theorems». Biology & Philosophy. 24 (4): 461–472. ISSN 0169-3867. PMC 2837227
 . PMID 20234823. doi:10.1007/s10539-008-9134-x. Consultado em 18 de novembro de 2018
. PMID 20234823. doi:10.1007/s10539-008-9134-x. Consultado em 18 de novembro de 2018 -
↑ Axe, Douglas D.; Dixon, Brendan W.; Lu, Philip (4 de junho de 2008). «Stylus: A System for Evolutionary Experimentation Based on a Protein/Proteome Model with Non-Arbitrary Functional Constraints». PLoS ONE (em inglês). 3 (6): e2246. ISSN 1932-6203. PMC 2405935
 . PMID 18523658. doi:10.1371/journal.pone.0002246
. PMID 18523658. doi:10.1371/journal.pone.0002246 -
↑ «Model and Laboratory Demonstrations That Evolutionary Optimization Works Well Only If Preceded by Invention--Selection Itself Is Not Inventive». BIO-Complexity (em inglês). 2015 (0). 22 de dezembro de 2015. ISSN 2151-7444
-
↑ «In the Beginning: Compelling Evidence for Creation and the Flood - References and Notes»
-
↑ Neto, Sodré Gonçalves de Brito. «Speciation in real time and historical-archaeological and its absence in geological time». Academia Journal of Scientific Research (em inglês)
-
↑ Neto, Sodré GB (12 de maio de 2017). «Speciation in real time and historical-archaeological and its absence in geological time DOI: 10.15413 / ajsr.2017.0413» Especiação em Tempo Real, em Tempo Arqueológico e sua Ausência em Tempo Geológico. Academia Publishing. Academia Journal Scientific Research journal. 7. ISSN 2315-7712. Consultado em 11 de abril de 2019 !CS1 manut: Data e ano (link)
-
↑ «Journal of Scientific Research: Covered By ISI, SCI: Impact Factor: 4.003»
-
↑ «Uma Dissensão Científica do Darwinismo – Dissent from Darwin». dissentfromdarwin.org. Consultado em 13 de julho de 2019
-
↑ «Galeria de Membros | TDI BRASIL»
-
↑ «Scopus preview - Scopus - Author details (Scherer, Siegfried)»
-
↑ «Scopus preview - Scopus - Author details (Bechly, Günter)»
-
↑ Bechly, Günter (13 de maio de 2020). «The Myth of Precambrian Sponges». Discovery Institute (em inglês). O Evolution News & Science Today (EN). Consultado em 13 de maio de 2020
-
↑ Lönnig, W. E.; Saedler, H. (31 de dezembro de 1997). «Plant transposons: contributors to evolution?». Gene. 205 (1-2): 245–253. ISSN 0378-1119. PMID 9461398
-
↑ Lönnig, Wolf-Ekkehard (2004). «http://www.sensortime.com/loennig-dygmosoic-e.htm». http://ressign.com/review_books_04/b_04/b04.htm 37/661 (2) ed. Kerala, Índia: Max-Planck-Institut for Plant Breeding Research, Carl-von-Linné-weg 10 . 50829 Cologne, Germany. ISBN 81-7736-231-3. Consultado em 7 de junho de 2019 Ligação externa em
|titulo=(ajuda) -
↑ Dawkins, Richard. (2001). O relojoeiro cego 1. reimpressão ed. São Paulo: Companhia das Letras. ISBN 8535901612. OCLC 124028651
-
↑ Park, Hyung Wook; Cho, Kyuhoon (2018). «Science, state, and spirituality: Stories of four creationists in South Korea». History of Science; an Annual Review of Literature, Research and Teaching. 56 (1): 35–71. ISSN 0073-2753. PMID 29241363. doi:10.1177/0073275317740268
-
↑ Behe, Michael J. (2009). «Waiting longer for two mutations». Genetics. 181 (2): 819–820; author reply 821–822. ISSN 0016-6731. PMC 2644969
 . PMID 19189948. doi:10.1534/genetics.108.098905
. PMID 19189948. doi:10.1534/genetics.108.098905 -
↑ Alvarez-Pereira, Carlos (2019). «Anticipations of Digital Sustainability: Self-Delusions, Disappointments and Expectations». Anticipation, Agency and Complexity. doi:10.1007/978-3-030-03623-2_7. Consultado em 11 de outubro de 2022
-
↑ Schubert, Mikkel; Jónsson, Hákon; Chang, Dan; Der Sarkissian, Clio; Ermini, Luca; Ginolhac, Aurélien; Albrechtsen, Anders; Dupanloup, Isabelle; Foucal, Adrien (30 de dezembro de 2014). «Prehistoric genomes reveal the genetic foundation and cost of horse domestication». Proceedings of the National Academy of Sciences (em inglês) (52). ISSN 0027-8424. PMC 4284583
 . PMID 25512547. doi:10.1073/pnas.1416991111. Consultado em 11 de outubro de 2022
. PMID 25512547. doi:10.1073/pnas.1416991111. Consultado em 11 de outubro de 2022 -
↑ Roizman, Bernard (1996). Baron, Samuel, ed. «Multiplication». Galveston (TX): University of Texas Medical Branch at Galveston. ISBN 978-0-9631172-1-2. PMID 21413311
-
↑ Bergman, Jerry (22 de abril de 1999). «Did God make pathogenic viruses? - creation.com» (em inglês). CMI has offices in Australia, Canada, Singapore, New Zealand, United Kingdom, South Africa and United States of America.: Journal of Creation. p. 13(1):115–125. Consultado em 11 de maio de 2020
-
↑ Basener, William F.; Sanford, John C. (1 de junho de 2018). «The fundamental theorem of natural selection with mutations». Journal of Mathematical Biology (em inglês). 76 (7): 1589–1622. ISSN 1432-1416. PMC 5906570
 . PMID 29116373. doi:10.1007/s00285-017-1190-x
. PMID 29116373. doi:10.1007/s00285-017-1190-x -
↑ Neto, Sodré (20 de dezembro de 2019). «As Múltiplas Funções do Apêndice » Portal TDI Brasil +». Portal TDI+. Consultado em 14 de maio de 220 Verifique data em:
|acessodata=(ajuda) -
↑ Armitage, Mark Hollis; Anderson, Kevin Lee (1 de julho de 2013). «Soft sheets of fibrillar bone from a fossil of the supraorbital horn of the dinosaur Triceratops horridus». Acta Histochemica (em inglês). 115 (6): 603–608. ISSN 0065-1281. doi:10.1016/j.acthis.2013.01.001
-
↑ Welch, John J. (1 de março de 2017). «What's wrong with evolutionary biology?». Biology & Philosophy (em inglês). 32 (2): 263–279. ISSN 1572-8404. PMC 5329086
 . PMID 28298744. doi:10.1007/s10539-016-9557-8
. PMID 28298744. doi:10.1007/s10539-016-9557-8 -
↑ «Published Reports of Original Soft Tissue Fossils». www.icr.org (em inglês). Consultado em 19 de maio de 2020
-
↑ «Radioisotopes and the Age of the Earth, Volume II». www.icr.org (em inglês). Consultado em 19 de maio de 2020
-
↑ «John Baumgardner». Wikipedia (em inglês). 25 de fevereiro de 2020
-
↑ Deactivation unit for radioactive materials, comprises capacitor with curved mantle surfaces, connected to controllable generator (em inglês), 20 de novembro de 2003, consultado em 11 de maio de 2020
-
↑ Deactivation unit for radioactive materials, comprises capacitor with curved mantle surfaces, connected to controllable generator (em inglês), 20 de novembro de 2003, consultado em 19 de maio de 2020
-
↑ Fan, Hui (1 de junho de 1995). «Decay rates in a piezoelectric strip». International Journal of Engineering Science (em inglês). 33 (8): 1095–1103. ISSN 0020-7225. doi:10.1016/0020-7225(94)00126-5
-
↑ «In the Beginning: Compelling Evidence for Creation and the Flood - The Origin of Earth's Radioactivity». www.creationscience.com. Consultado em 19 de maio de 2020
-
↑ Eiseley, Loren C. (1945). «Tempo and mode in evolution. By George Gaylord Simpson. Columbia University Press, New York City, 1944. 217 pp. of text, bibliography, and index. 36 figures, 19 tables. Price $3.50». American Journal of Physical Anthropology (em inglês). 3 (2): 208–209. ISSN 1096-8644. doi:10.1002/ajpa.1330030215
-
↑ Gould, Stephen Jay; Eldredge, Niles (1977). «Punctuated equilibria: the tempo and mode of evolution reconsidered». Paleobiology (em inglês). 3 (2): 115–151. ISSN 0094-8373. doi:10.1017/S0094837300005224
-
↑ Estes, Suzanne; Arnold, Stevan J. (1 de fevereiro de 2007). «Resolving the Paradox of Stasis: Models with Stabilizing Selection Explain Evolutionary Divergence on All Timescales.». The American Naturalist. 169 (2): 227–244. ISSN 0003-0147. doi:10.1086/510633
-
↑ Wake, David B.; Roth, Gerhard; Wake, Marvalee H. (21 de março de 1983). «On the problem of stasis in organismal evolution». Journal of Theoretical Biology (em inglês). 101 (2): 211–224. ISSN 0022-5193. doi:10.1016/0022-5193(83)90335-1
-
↑ Lönnig, Wolf-Ekkehard (2004). «Dynamic genomes, morphological stasis, and the origin of irreducible complexity». www.sensortime.com. Kerala, India.: Max-Planck-Institut for Plant Breeding Research, Carl-von-Linné-weg 10 50829 Cologne, Germany. pp. 37/661 (2). ISBN 81-7736-231-3. Consultado em 19 de maio de 2020
-
↑ Goodfellow, Wayne D.; Nowlan, Godfrey S.; McCracken, Alexander D.; Lenz, Alfred C.; Grégoire, D. Conrad (1 de janeiro de 1992). «Geochemical anomalies near the Ordovician‐Silurian boundary, Northern Yukon Territory, Canada». Historical Biology (1): 1–23. ISSN 0891-2963. doi:10.1080/10292389209380415. Consultado em 11 de abril de 2022
-
↑ Furness, Andrew I.; Lee, Kevin; Reznick, David N. (junho de 2015). «Adaptation in a variable environment: Phenotypic plasticity and bet-hedging during egg diapause and hatching in an annual killifish». Evolution; International Journal of Organic Evolution. 69 (6): 1461–1475. ISSN 1558-5646. PMID 25908306. doi:10.1111/evo.12669
-
↑ Keim, Brandon. «Birth of New Species Witnessed by Scientists». WIRED (em inglês)
-
↑ «Speciation in real time». evolution.berkeley.edu. Consultado em 10 de agosto de 2017
-
↑ Gerke, Justin (8 de novembro de 2008). «Speciation in Real-Time». Science 2.0 (em inglês). Consultado em 10 de agosto de 2017
-
↑ Reichenbacher, Bettina; Sienknecht, Ulrike; Küchenhoff, Helmut; Fenske, Nora (outubro de 2007). «Combined otolith morphology and morphometry for assessing taxonomy and diversity in fossil and extant killifish (Aphanius, Prolebias)». Journal of Morphology. 268 (10): 898–915. ISSN 0362-2525. PMID 17674357. doi:10.1002/jmor.10561
-
↑ «Mechanisms: the processes of evolution». evolution.berkeley.edu. Consultado em 10 de agosto de 2017
-
↑ Varón-González, Ceferino; Whelan, Simon; Klingenberg, Peter. «Estimating Phylogenies from Shape and Similar Multidimensional Data: Why It Is Not Reliable». Systematic Biology (em inglês). doi:10.1093/sysbio/syaa003
-
↑ Varón-González, Ceferino; Whelan, Simon; Klingenberg, Peter. «Estimating Phylogenies from Shape and Similar Multidimensional Data: Why It Is Not Reliable». Systematic Biology (em inglês). doi:10.1093/sysbio/syaa003
-
↑ Peretó, Juli; Bada, Jeffrey L.; Lazcano, Antonio (1 de outubro de 2009). «Charles Darwin and the Origin of Life». Origins of Life and Evolution of Biospheres (em inglês). 39 (5): 395–406. ISSN 1573-0875. PMC 2745620
 . PMID 19633921. doi:10.1007/s11084-009-9172-7
. PMID 19633921. doi:10.1007/s11084-009-9172-7 -
↑ Scherer, Siegfried (1 de agosto de 1985). «Could life have arisen in the primitive atmosphere?». Journal of Molecular Evolution (em inglês) (1): 91–94. ISSN 1432-1432. doi:10.1007/BF02105809. Consultado em 11 de abril de 2022
-
↑ Pantaloni, Marco; Console, Fabiana; Lorusso, Lorenzo; Petti, Fabio Massimo; Franchini, Antonia Francesca; Porro, Alessandro; Romano, Marco (1 de janeiro de 2017). «Italian physicians' contribution to geosciences». Geological Society, London, Special Publications (em inglês). 452 (1): 55–75. ISSN 0305-8719. doi:10.1144/SP452.17
-
↑ «The Geological Society of London - Wollaston Medal». www.geolsoc.org.uk. Consultado em 10 de agosto de 2017
-
↑ Faria, Frederico Felipe de Almeida (2010). Georges Cuvier e a instauração da Paleontologia como ciência (PDF). Florianópolis, SC: Tese de doutorado - UFSC
-
↑ Rudwick, Martin (2005). Bursting the Limits of Time: The Reconstruction of Geohistory in the Age of Revolution, 2005 - The University of Chicago Press. [S.l.: s.n.] ISBN 0-226-73111-1
-
↑ Williamson, Peter G. (19 de novembro de 1981). «Morphological stasis and developmental constraint: real problems for neo-Darwinism». Nature (em inglês). 294 (5838): 214–215. doi:10.1038/294214a0
-
↑ Davis, Charles C.; Schaefer, Hanno; Xi, Zhenxiang; Baum, David A.; Donoghue, Michael J.; Harmon, Luke J. (22 de abril de 2014). «Long-term morphological stasis maintained by a plant–pollinator mutualism». Proceedings of the National Academy of Sciences of the United States of America. 111 (16): 5914–5919. ISSN 0027-8424. PMID 24706921. doi:10.1073/pnas.1403157111
-
↑ Lönnig, Wolf-Ekkehard (2005). «Dynamic genomes, morphological stasis, and the origin of irreducible complexity». Max-Planck-Institut for Plant Breeding Research. Research Signpost 37/661 (2), Fort P.O., Trivandrum-695 023, Kerala, India .Dynamical Genetics, 2004: 101-119: internetlibrary.html. ISBN 81-7736-231-3. Consultado em 1 de fevereiro de 2018
-
↑ Welch, John J. (1 de março de 2017). «What's wrong with evolutionary biology?». Biology & Philosophy (em inglês). 32 (2): 263–279. ISSN 0169-3867. doi:10.1007/s10539-016-9557-8
-
↑ John, Whitmore, (2013). «Temporal Patterns in 'Living Fossils'» (em inglês)
-
↑ Ghalambor, Cameron K.; Hoke, Kim L.; Ruell, Emily W.; Fischer, Eva K.; Reznick, David N.; Hughes, Kimberly A. (17 de setembro de 2015). «Non-adaptive plasticity potentiates rapid adaptive evolution of gene expression in nature». Nature (em inglês). 525 (7569): 372–375. doi:10.1038/nature15256
-
↑ Ezard, Thomas H. G.; Quental, Tiago B.; Benton, Michael J. (5 de abril de 2016). «The challenges to inferring the regulators of biodiversity in deep time». Phil. Trans. R. Soc. B (em inglês). 371 (1691). 20150216 páginas. ISSN 0962-8436. PMID 26977058. doi:10.1098/rstb.2015.0216
-
↑ McKinney, Michael L. (1 de novembro de 1986). «How Biostratigraphic Gaps Form». The Journal of Geology. 94 (6): 875–884. ISSN 0022-1376. doi:10.1086/629093
-
↑ McGill, Brian J.; Dornelas, Maria; Gotelli, Nicholas J.; Magurran, Anne E. (1 de fevereiro de 2015). «Fifteen forms of biodiversity trend in the Anthropocene». Trends in Ecology & Evolution (em English). 30 (2): 104–113. ISSN 0169-5347. PMID 25542312. doi:10.1016/j.tree.2014.11.006 !CS1 manut: Língua não reconhecida (link)
-
↑ Grant, B. Rosemary; Grant, Peter R. (3 de março de 2017). «Watching speciation in action». Science (New York, N.Y.). 355 (6328): 910–911. ISSN 1095-9203. PMID 28254901. doi:10.1126/science.aam6411
-
↑ Gera, Dinesh; Syamlal, Madhava; O'Brien, Thomas J. (1 de abril de 2004). «Hydrodynamics of particle segregation in fluidized beds». International Journal of Multiphase Flow. 30 (4): 419–428. doi:10.1016/j.ijmultiphaseflow.2004.01.003
-
↑ Meakin, Paul (1 de março de 1990). «A simple two-dimensional model for particle segregation». Physica A: Statistical Mechanics and its Applications. 163 (3): 733–746. doi:10.1016/0378-4371(90)90247-P
-
↑ Makse, Hernán A.; Havlin, Shlomo; King, Peter R.; Stanley, H. Eugene (27 de março de 1997). «Spontaneous stratification in granular mixtures». Nature (em inglês). 386 (6623): 379–382. doi:10.1038/386379a0
-
↑ Alden, Andrew. «Steno Started Geology With a Few Simple Principles». ThoughtCo
-
↑ Martini, I. Peter; Baker, Victor R.; Garzón, Guillermina (5 de março de 2009). Flood and Megaflood Processes and Deposits: Recent and Ancient Examples (Special Publication 32 of the IAS) (em inglês). [S.l.]: John Wiley & Sons. ISBN 9781444304305
-
↑ Martini, I. Peter; Baker, Victor R.; Garzón, Guillermina (5 de março de 2009). Flood and Megaflood Processes and Deposits: Recent and Ancient Examples (Special Publication 32 of the IAS) (em inglês). [S.l.]: John Wiley & Sons. ISBN 9781444304305
-
↑ Berthault, G.; Lalomov, A. V.; Tugarova, M. A. (1 de janeiro de 2011). «Reconstruction of paleolithodynamic formation conditions of Cambrian-Ordovician sandstones in the Northwestern Russian platform». Lithology and Mineral Resources (em inglês). 46 (1): 60–70. ISSN 0024-4902. doi:10.1134/S0024490211010020
-
↑ Ormö, Jens; Sturkell, Erik; Alwmark, Carl; Melosh, Jay (23 de outubro de 2014). «First known Terrestrial Impact of a Binary Asteroid from a Main Belt Breakup Event». Scientific Reports (em inglês). 4 (1). ISSN 2045-2322. doi:10.1038/srep06724
-
↑ «Figure 2: Paleogeography of Baltica and neighboring cratons at the time of the increased cosmic bombardment following the ~470 Ma asteroid breakup event, and timeline for the related meteorite falls (black dot and line) as well as known craters (red dots).». www.nature.com (em inglês). Consultado em 8 de janeiro de 2018
-
↑ Hassler, Scott W.; Simonson, Bruce M. (2001). «The Sedimentary Record of Extraterrestrial Impacts in Deep‐Shelf Environments: Evidence from the Early Precambrian». The Journal of Geology. 109 (1): 1–19. doi:10.1086/317958
-
↑ Glikson, A.Y.; Allen, C.; Vickers, J. (2004). «Multiple 3.47-Ga-old asteroid impact fallout units, Pilbara Craton, Western Australia☆». Earth and Planetary Science Letters. 221 (1-4): 383–396. doi:10.1016/s0012-821x(04)00104-9. Consultado em 3 de fevereiro de 2018
-
↑ Heck, Philipp R.; Schmitz, Birger; Bottke, William F.; Rout, Surya S.; Kita, Noriko T.; Cronholm, Anders; Defouilloy, Céline; Dronov, Andrei; Terfelt, Fredrik (fevereiro de 2017). «Rare meteorites common in the Ordovician period». Nature Astronomy (em inglês). 1 (2). ISSN 2397-3366. doi:10.1038/s41550-016-0035
-
↑ Bunch, Ted E.; Hermes, Robert E.; Moore, Andrew M.T.; Kennett, Douglas J.; Weaver, James C.; Wittke, James H.; DeCarli, Paul S.; Bischoff, James L.; Hillman, Gordon C. (10 de julho de 2012). «Very high-temperature impact melt products as evidence for cosmic airbursts and impacts 12,900 years ago». Proceedings of the National Academy of Sciences of the United States of America. 109 (28): E1903–E1912. ISSN 0027-8424. PMID 22711809. doi:10.1073/pnas.1204453109
-
↑ Wei, Young (15 de maio de 2014). «Oxygen escape from the Earth during geomagnetic reversals: Implications to mass extinction». Earth and Planetary Science Letters (em inglês). 394: 94–98. ISSN 0012-821X. doi:10.1016/j.epsl.2014.03.018
-
↑ Sleep, Norman H.; Lowe, Donald R. (1 de abril de 2014). «Physics of crustal fracturing and chert dike formation triggered by asteroid impact, ∼3.26 Ga, Barberton greenstone belt, South Africa». Geochemistry, Geophysics, Geosystems (em inglês). 15 (4): 1054–1070. ISSN 1525-2027. doi:10.1002/2014gc005229
-
↑ Maruyama, Shigenori; Santosh, M.; Azuma, Shintaro. «Initiation of plate tectonics in the Hadean: Eclogitization triggered by the ABEL Bombardment». Geoscience Frontiers. doi:10.1016/j.gsf.2016.11.009
-
↑ Maruyama, Shigenori; Ebisuzaki, Toshikazu. «Origin of the Earth: A proposal of new model called ABEL». Geoscience Frontiers. 8 (2): 253–274. doi:10.1016/j.gsf.2016.10.005
-
↑ Lönnig, Wolf-Ekkehard (2004). «Dynamic genomes, morphological stasis, and the origin of irreducible complexity». www.sensortime.com. Max-Planck-Institut for Plant Breeding Research. Consultado em 20 de março de 2021
-
↑ Crabtree, Gerald R. (janeiro de 2013). «Our fragile intellect. Part II». Trends in genetics: TIG (1): 3–5. ISSN 0168-9525. PMID 23153597. doi:10.1016/j.tig.2012.10.003. Consultado em 11 de outubro de 2022
-
↑ Gorelick*, Root (2005). «Environmentally alterable additive genetic effects». Evolutionary Ecology Research (em English). 7 (3). ISSN 1522-0613 !CS1 manut: Língua não reconhecida (link)
-
↑ Furness, Andrew I.; Lee, Kevin; Reznick, David N. (junho de 2015). «Adaptation in a variable environment: Phenotypic plasticity and bet-hedging during egg diapause and hatching in an annual killifish». Evolution; International Journal of Organic Evolution. 69 (6): 1461–1475. ISSN 1558-5646. PMID 25908306. doi:10.1111/evo.12669
-
↑ Keim, Brandon. «Birth of New Species Witnessed by Scientists». WIRED (em inglês)
-
↑ «Speciation in real time». evolution.berkeley.edu. Consultado em 10 de agosto de 2017
-
↑ Gerke, Justin (8 de novembro de 2008). «Speciation in Real-Time». Science 2.0 (em inglês). Consultado em 10 de agosto de 2017
-
↑ Reichenbacher, Bettina; Sienknecht, Ulrike; Küchenhoff, Helmut; Fenske, Nora (outubro de 2007). «Combined otolith morphology and morphometry for assessing taxonomy and diversity in fossil and extant killifish (Aphanius, Prolebias)». Journal of Morphology. 268 (10): 898–915. ISSN 0362-2525. PMID 17674357. doi:10.1002/jmor.10561
-
↑ «Mechanisms: the processes of evolution». evolution.berkeley.edu. Consultado em 10 de agosto de 2017
-
↑ Salisbury, Annis (14 de outubro de 1916). «The Tell-Tale Fossil». Scientific American (16): 348–349. ISSN 0036-8733. doi:10.1038/scientificamerican10141916-348. Consultado em 11 de abril de 2022
-
↑ Park, Hyung Wook; Cho, Kyuhoon (2018). «Science, state, and spirituality: Stories of four creationists in South Korea». History of Science; an Annual Review of Literature, Research and Teaching. 56 (1): 35–71. ISSN 0073-2753. PMID 29241363. doi:10.1177/0073275317740268
-
↑ Groto, Sílvia Regina; Martins, André Ferrer P. (2018). Prestes, Maria Elice de Brzezinski; Silva, Cibelle Celestino, eds. «The "Science" as Portrayed in Documents of the Biological Evolution Versus Intelligent Design Debate». Cham: Springer International Publishing. Science: Philosophy, History and Education (em inglês): 125–141. ISBN 9783319740362. doi:10.1007/978-3-319-74036-2_9
-
↑ Blancke, Stefaan; De Smedt, Johan; De Cruz, Helen; Boudry, Maarten; Braeckman, Johan (1 de agosto de 2012). «The Implications of the Cognitive Sciences for the Relation Between Religion and Science Education: The Case of Evolutionary Theory». Science & Education (em inglês). 21 (8): 1167–1184. ISSN 1573-1901. doi:10.1007/s11191-011-9402-z
-
↑ Adami, Christoph; Ofria, Charles; Collier, Travis C. (25 de abril de 2000). «Evolution of biological complexity». Proceedings of the National Academy of Sciences (em inglês). 97 (9): 4463–4468. ISSN 0027-8424. PMID 10781045. doi:10.1073/pnas.97.9.4463
-
↑ Basener, William F.; Sanford, John C. (1 de junho de 2018). «The fundamental theorem of natural selection with mutations». Journal of Mathematical Biology (em inglês) (7): 1589–1622. ISSN 1432-1416. PMC 5906570
 . PMID 29116373. doi:10.1007/s00285-017-1190-x. Consultado em 11 de abril de 2022
. PMID 29116373. doi:10.1007/s00285-017-1190-x. Consultado em 11 de abril de 2022 -
↑ Smith, Thomas; Ho, Gladys; Christodoulou, John; Price, Elizabeth Ann; Onadim, Zerrin; Gauthier-Villars, Marion; Dehainault, Catherine; Houdayer, Claude; Parfait, Beatrice (maio de 2016). «Extensive Variation in the Mutation Rate Between and Within Human Genes Associated with Mendelian Disease: HUMAN MUTATION». Human Mutation (em inglês) (5): 488–494. doi:10.1002/humu.22967. Consultado em 17 de dezembro de 2022
-
↑ Berthault, G.; Lalomov, A. V.; Tugarova, M. A. (1 de janeiro de 2011). «Reconstruction of paleolithodynamic formation conditions of Cambrian-Ordovician sandstones in the Northwestern Russian platform». Lithology and Mineral Resources (em inglês) (1): 60–70. ISSN 1608-3229. doi:10.1134/S0024490211010020. Consultado em 11 de abril de 2022
-
1 2 Sodré, GBN (9 de setembro de 2021). «Como o Criacionismo Explica o Registro fóssil, as Camadas Geológicas Sedimentares e a Repetição do mesmo Número de "Templates" Fósseis (TF) se Mantendo até hoje mesmo sob Número Gigante de milhões de Espécies na Biodiversidade Atual?». Jornal da Ciência. doi:10.13140/RG.2.2.32724.12168. Consultado em 11 de abril de 2022
-
1 2 Sodré, GBN (23 de setembro de 2021). «Templates Fósseis (TF) e Templates Biológicos (TB) Permanecem Praticamente Iguais». Jornal da Ciência. doi:10.13140/RG.2.2.26021.17124. Consultado em 12 de abril de 2022
-
1 2 Guy Berthault, Guy (2013). «Orogenesis: Cause of Sedimentary Formations». Open Journal of Geology (02): 22–24. ISSN 2161-7570. doi:10.4236/ojg.2013.32B005. Consultado em 12 de abril de 2022
-
↑ Neto, Sodré Gonçalves de Brito (1 de janeiro de 2017). «Speciation in real time and historical-archaeological and its absence in geological time». Academia Journal of Scientific Research. doi:10.15413/ajsr.2017.0413. Consultado em 11 de abril de 2022