Autor: Sodré G. B. Neto
Afiliação: IPPTM – Instituto de Pesquisa em Paleogenética, TP53 e MicroRNA / CEGH / ICB / UFG
Resumo:
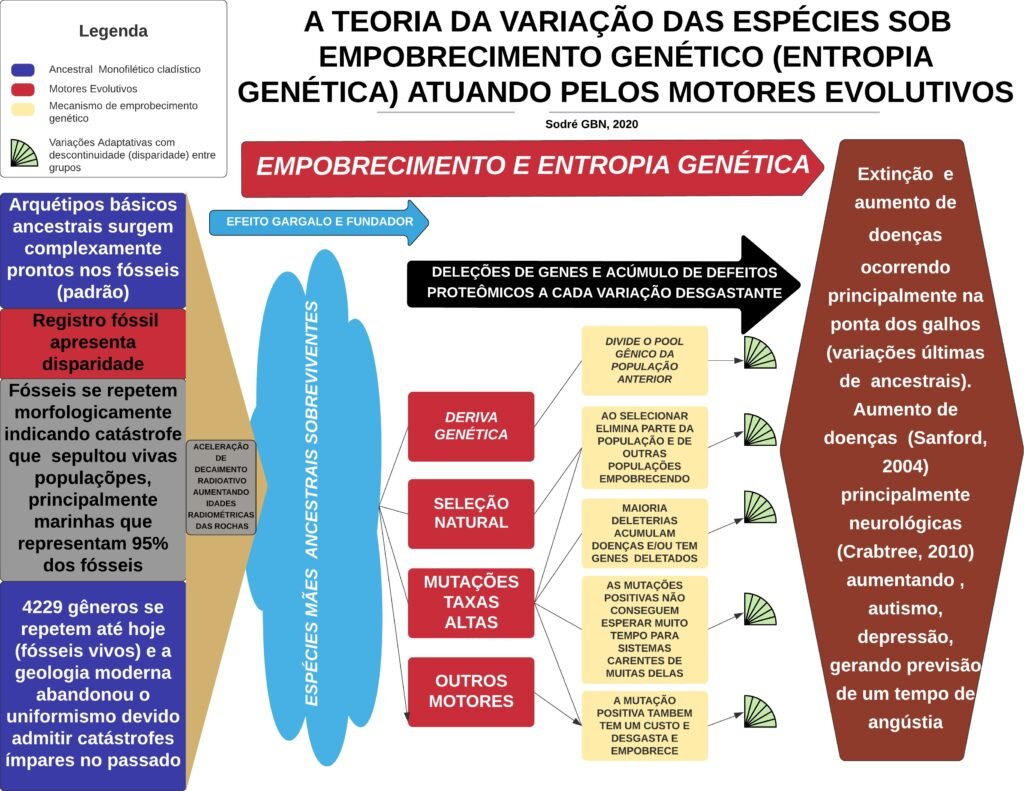
Introdução: A teoria da evolução biológica convencional fundamenta-se na premissa de que mutações aleatórias e seleção natural são capazes de gerar complexidade e viabilidade a longo prazo. No entanto, avanços na genética de populações e genômica comparativa sugerem um processo inverso: a degeneração sistemática do genoma pelo acúmulo de mutações deletérias, um fenômeno descrito como entropia genética [299].
Objetivo: Este artigo sintetiza evidências sobre o acúmulo de carga genética deletéria na linhagem humana e animal, propondo a ocorrência de um pico mutacional/radiativo recente durante o Holoceno, desencadeado por eventos catastróficos.
Métodos: Realizou-se uma revisão sistemática de literatura focada em taxas de mutação mitocondrial (mtDNA), acúmulo de mutações deletérias em populações pequenas e grandes, e o impacto de eventos catastróficos na geocronologia radioativa.
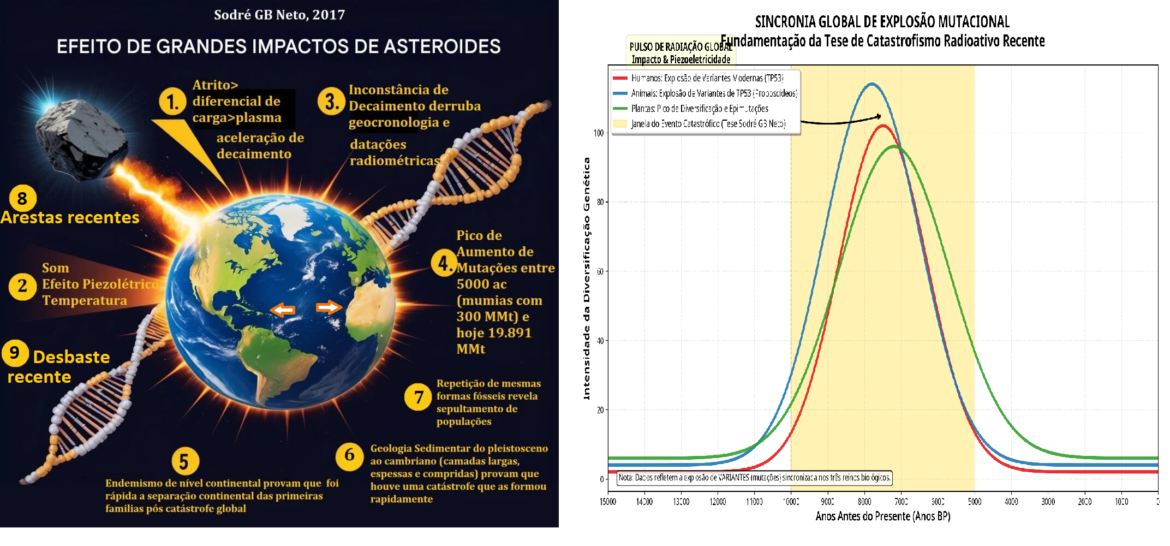
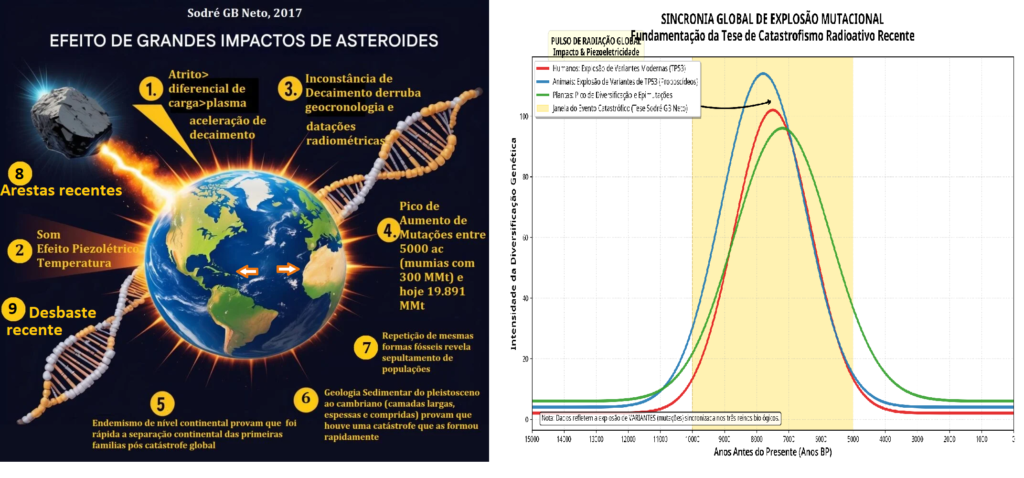
Resultados: As taxas de mutação observadas em estudos de linhagem (pedigree) são significativamente superiores às taxas filogenéticas tradicionais, indicando um erro sistemático na calibração do relógio molecular. Evidências geológicas sugerem que impactos de asteroides podem ter induzido picos de radiação via piezoeletricidade nuclear, acelerando o decaimento radioativo e as taxas mutacionais entre 5.000 e 10.000 anos atrás. A perda de funcionalidade em genes críticos, como o supressor tumoral p53, reflete essa senescência evolutiva.
Conclusão: A trajetória atual de acúmulo de mutações (deleteriome) aponta para um “mutational meltdown”, sugerindo que a viabilidade das espécies contemporâneas está severamente comprometida, com uma perspectiva de extinção iminente em termos geológicos.
Introdução
Métodos
Resultados
O Pico Mutacional Holocênico
Catastrofismo Radioativo e Geocronologia
Senescência Evolutiva e o Deleteriome
Discussão
Conclusão
Referências
302.https://pmc.ncbi.nlm.nih.gov/articles/PMC10993466/
303. Markova, A. K., & van Kolfschoten, T. (2008). The mammal fauna in the Late Pleistocene and Holocene of the East European plain. Quaternary International, 179(1), 69-79.
304. Walker, M., et al. (2019). Formal definition and dating of the GSSP (Global Stratotype Section and Point) for the base of the Holocene using the Greenland NGRIP ice core, and selected auxiliary records. Journal of Quaternary Science, 34(3), 1-18.
305. Birks, H. J. B., & Birks, H. H. (2004). The rise and fall of forests. Science, 305(5683), 484-485.
306. Domínguez-Vázquez, G., et al. (2020). Changes in vegetation structure during the Pleistocene-Holocene transition in Guanajuato, Central México. Review of Palaeobotany and Palynology, 279, 104245.
307. Fedoroff, N. V., et al. (2010). Radically rethinking agriculture for the 21st century. Science, 327(5967), 833-834. (PMID: 20150485)
308. Rull, V. (2011). Neotropical biodiversity and conservation in the end-Cenozoic icehouse world. Progress in Physical Geography, 35(5), 579-604.
309. Hadly, E. A., et al. (2004). Genetic response to climatic change: insights from ancient DNA and phylochronology. PLoS Biology, 2(10), e290. (PMID: 15383847, PMCID: PMC514476)
310. Gill, R. A., et al. (2015). The role of DNA repair in the plant response to abiotic stress. Genes, 6(3), 899-922. (PMID: 26404349, PMCID: PMC4598659)
311. Roldán-Arjona, T., & Ariza, R. R. (2009). DNA base excision repair in plants. Advances in Botanical Research, 50, 183-217.
312. Hartmann, A., et al. (2021). Stress Management in Plants: Examining Provisional and Unique Dose-Dependent Responses. International Journal of Molecular Sciences, 22(16), 8899. (PMID: 34445662, PMCID: PMC8395579)
313. Sandler, G. A., et al. (2020). Estimation of the SNP Mutation Rate in Two Vegetatively Propagating Species of Duckweed. G3: Genes, Genomes, Genetics, 10(11), 4017-4024. (PMID: 32973000, PMCID: PMC7642947)
314. Jiang, C., et al. (2014). The effects of thermal stress on the rates of molecular evolution in the plant genus Arabidopsis. Molecular Ecology, 23(8), 1953-1965. (PMID: 24612218)
315. Zhu, J. K. (2016). Abiotic stress signaling and responses in plants. Cell, 167(2), 313-324. (PMID: 27716505, PMCID: PMC5104236)
316. Juliana, P., et al. (2017). Natural genetic resources from diverse plants to improve abiotic stress tolerance in plants. International Journal of Molecular Sciences, 18(10), 2049. (PMID: 28934131, PMCID: PMC5666759)
317. Ito, H. (2022). Environmental stress and transposons in plants. Genes & Genetic Systems, 97(4), 169-175. (PMID: 35922238)
318. Grandbastien, M. A. (2015). Stress-induced activation of transposable elements in plants. Current Opinion in Plant Biology, 27, 74-81. (PMID: 26115369)
319. Cavrak, V. V., et al. (2014). How a retrotransposon exploits the plant’s heat stress response for its activation. PLoS Genetics, 10(1), e1004115. (PMID: 24497843, PMCID: PMC3907370)
320. Willerslev, E., et al. (2014). Fifty thousand years of Arctic vegetation and megafaunal diet. Nature, 506(7486), 47-51. (PMID: 24499916, PMCID: PMC4019438)
321. Giguet-Covex, C., et al. (2014). Long-term trajectories of biodiversity in a mountain lake ecosystem. Nature Communications, 5, 3234. (PMID: 24513718)
322. Alsos, I. G., et al. (2020). High resolution ancient sedimentary DNA shows that alpine plant diversity is associated with human land use and climate change. Nature Communications, 11(1), 5536. (PMID: 33149118, PMCID: PMC7642359)
323. Schultz, S. T., & Scofield, D. G. (2019). Somatic Mutation and Evolution in Plants. Annual Review of Ecology, Evolution, and Systematics, 50, 49-73.
324. Lesaffre, T., et al. (2021). Population-level consequences of inheritable somatic mutations and the evolution of mutation rates in plants. eLife, 10, e68535. (PMID: 34491218, PMCID: PMC8435380)
325. Watson, J. M., et al. (2016). The genomic scale of fluctuating selection in a natural plant population. Genetics, 202(2), 721-735. (PMID: 26675512, PMCID: PMC4751564)
326. Manova, V., & Grusz, A. (2015). DNA damage and repair in plants – from models to crops. Frontiers in Plant Science, 6, 885. (PMID: 26579163, PMCID: PMC4633482)
327. Waterworth, M. R., et al. (2011). The plant DNA damage response. Biochimica et Biophysica Acta (BBA) – Gene Regulatory Mechanisms, 1819(7), 776-787.
328. Schemske, D. W., et al. (2010). Is there a latitudinal gradient in the importance of biotic interactions? Annual Review of Ecology, Evolution, and Systematics, 41, 245-269.
329. Macovei, A., & Tuteja, N. (2022). The Role of DNA Repair in the Plant Response to Abiotic Stress. Genes, 13(5), 899. (PMID: 35627284, PMCID: PMC9141019)
330. Frankham, R. (2005). Genetics and extinction. Biological Conservation, 126(2), 131-140.
331. Fu, Q., et al. (2014). A revised timescale for human evolution based on ancient mitochondrial genomes. Current Biology, 24(5), 553-559. (PMID: 24582340, PMCID: PMC3950946)
332. Muscheler, R., et al. (2007). ¹⁰Be and ¹⁴C in the Greenland Ice Core Project ice core: reconstruction of the solar activity and its relation to the climate. Quaternary Science Reviews, 26(1-2), 82-97.
333. Miyake, F., et al. (2012). A signature of cosmic-ray increase in AD 774–775 from tree rings in Japan. Nature, 486(7402), 240-242. (PMID: 22699615)
334. Cooper, A., et al. (2021). A global environmental crisis 42,000 years ago. Science, 371(6531), 811-818. (PMID: 33602851)
335. Laj, C., & Kissel, C. (2015). The Laschamp-Mono Lake geomagnetic excursions: A new look from the North Atlantic. Geophysical Research Letters, 42(16), 6605-6613.